
无垠的微观世界
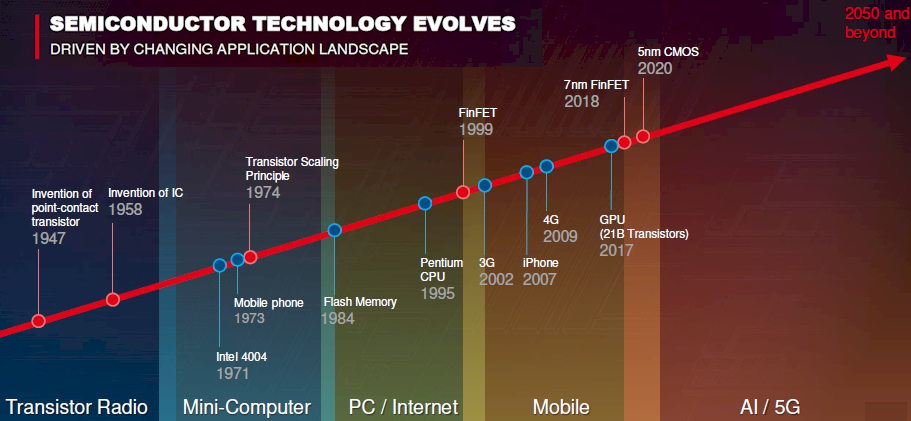
1959年的冬天,物理学家Richard Feynman在加州理工举办的美国物理年会上作了题为"There's Plenty of Room at the Bottom: An Invitation to Enter a New Field of Physics"的演讲,在这个轰动世界的演讲中,他向人们描述了一个奇幻的世界。在这个世界中,大英百科全书被记录在大头针的针头上;人们可以通过电子显微镜看到一个原子和DNA上突变的碱基;大量的信息可以存储在很小的空间里,字母可以用六或七位的字符表示..。这些预言在当代已经实现,他当时甚至还提出了要将计算机和晶体管等电子器件做的更小,还阐述了像光刻和蒸镀这种现代芯片的工艺技术的思路。
巧合的是,演讲的同一年,Mohamed Atalla和Dawon Kahng在远在东海岸的贝尔实验室中发明了金属氧化物半导体场效应晶体管(MOSFET),这是真正意义上的第一个可以微型化且批量生产的晶体管。从那年开始,晶体管被做的更小,更多的晶体管被集成在一个电路上。

1965年的春天,也就是Feynman演讲的6年之后,Fairchild Semiconductor和Intel的创始人,同在加州的Gordon Moore,提出了一个惊人的观点,他认为集成电路中晶体管的密度每年都会翻倍,晶体管的体积每年缩小一倍,并且这种速度会持续至少十年,十年后他将这个速度修正为每18-24个月,他修正后的观点神奇地预测了历史,即为著名的摩尔定律。在摩尔定律自我预言实现的驱使下,集成电路微处理器的性能不断翻倍,且伴随着价格的快速下降,晶体管的尺寸下降到了微米甚至纳米级。1988年秋天,一个名为“分子电子器件”的特殊会议召开了,在这一年的早些时候,Aviram详细的描述了一个理论上摩尔定律下晶体管微型化的最终目标 – 单分子晶体管,因为单个分子构成了可想象中的最小的稳定结构,在这个会议上,科学家们提出了各种各样的单分子所构成的电子器件的设想,讨论着摩尔定律未来的终局。可以说,摩尔定律指导了人类在半导体行业的长期规划和研发目标的设定,在我们的生物科技领域,摩尔定律也同样指导了一个无比重要的研究方向 – 下一代测序技术(next-generation sequencing),也就是我们常说的NGS或二代测序技术。
理想中的单分子测序
同曼哈顿原子弹计划和阿波罗计划并称为三大科学计划的人类基因组计划(Human Genome Project)于1990年在全世界范围内展开了,它被誉为生命科学的“登月计划”,历时十三年耗资30亿美金,测序工作的完成解开了人体内2.5万个基因的30亿个碱基对的秘密,但当我们把图谱和花费联系到一起时,发现平均检测1个碱基需要1美元,这个成本成为了个体化常规化基因组测序不可逾越的鸿沟。

在人类基因组计划接近尾声的时候,科学家们开始讨论如何将基因组测序的时间缩短,成本降低。2002年波士顿的GSAC会议(The Genome Sequencing and Analysis Conference)的开幕演讲中,包括George M. Church等基因测序的先驱们发表了名为“The Future of Sequencing: Advancing Towards the $1000 Genome.”的演讲,这一演讲明确了人类向1000美元测序时代冲刺的决心,两年后美国人类基因组研究所(National Human Genome Research Institute)提出了推动突破性测序技术发展的特殊计划,我们很有幸也参与其中。
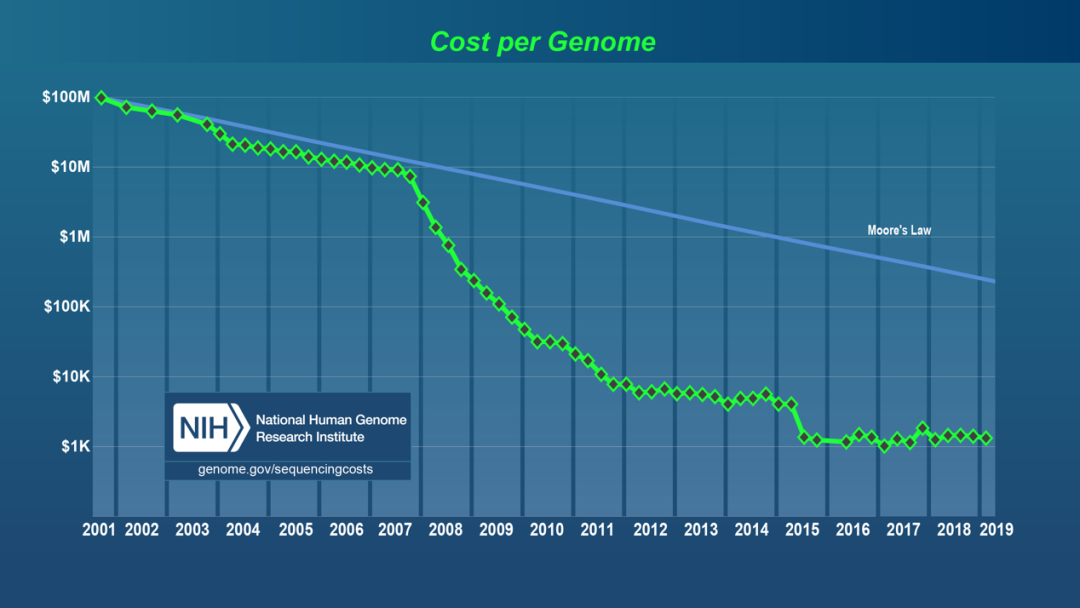
当时参与这个计划的几个团队都不约而同的想到了同一个方向 – 单分子测序,同单分子晶体管一样,对单个核酸分子测序是可想象的最小成本、最短时间的测序方案,以往的测序方案都需要做PCR扩增,且重复地测同一个序列,在当时的实验能力与原材料产能不足的情况下,测序的成本很难被降低。在单分子测序的方向中,产生了几个著名的商业化公司,也都采用了不同的很有趣的测序方法。
Helicos Biosciences
Helicos的创始人之一Stephen Quake,是瀚海基因创始人贺建奎博士做博士后时的导师,公司的Helicos Genetic Analysis Platform是第一个通过对单个DNA分子成像来实现基因测序技术的仪器。Helicos也是采用边合成边测序(Sequencing by Synthesis)的方法,预先将多个单分子单链DNA固定在基底上,每次只添加一种有荧光标记的单核苷酸,通过聚合酶进行合成,在反应后直接显微成像,有荧光标记的分子被记录为完成了一个碱基的配对,不断地重复AGCT四种核苷酸的配对合成直到完成整条单链DNA的配对,如右侧图下面的表格所示,对记录的三条单链DNA分子不断重复后直接读出了连续的15个碱基序列。
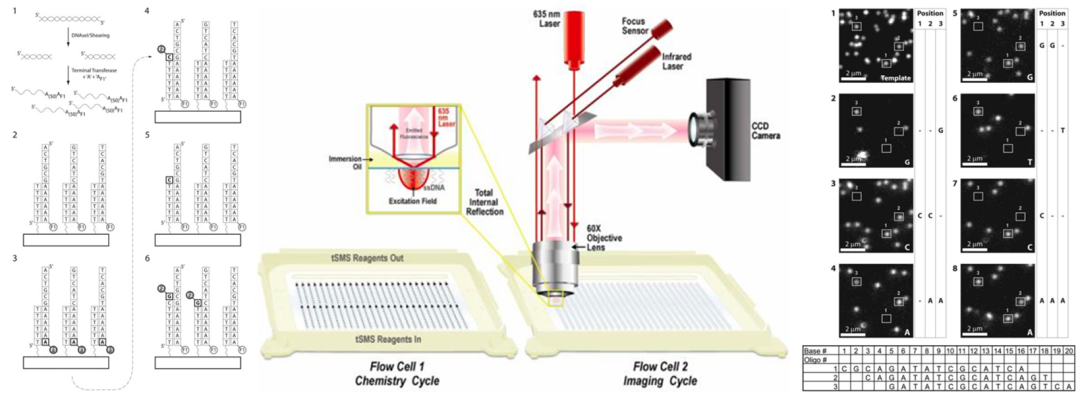
在Helicos的设备中,一个关键技术是中间图所示的全内反射荧光(total internal reflection fluorescence)成像,因为麦克斯韦方程组对光(电磁波)的描述为连续方程,当光在两个不同折射率所形成的界面之间反射的时候,会沿着界面产生一个光的渐逝场,场强随着离开界面的距离指数递减,非常灵敏,且场的作用距离只有光波长的几分之一,将背景中的噪音降到最低,在当时Helicos商业化的设备中以这种结束通过物镜对样本的扫描可以在很大面积的基底上进行高灵敏度的单分子荧光成像,从而完成单分子测序。

Pacific Biosciences
2003年1月的Science杂志发表了一篇名为“Zero-Mode Waveguides for Single-Molecule Analysis at High Concentrations”的封面文章,文章中描述了一种可高平行同时检测单分子的动态过程的方法,这个装置实现方法非常简单,即在熔融石英表面上的金属铝镀膜中做出数十纳米的微孔的阵列,由于孔的直径小于入射光的波长,入射光主要的能量被限制在微孔的底部,无法传播出去,因此被称为零模波导,这种芯片器件能以微秒的时间分辨率研究微摩尔浓度下的单分子动力学。
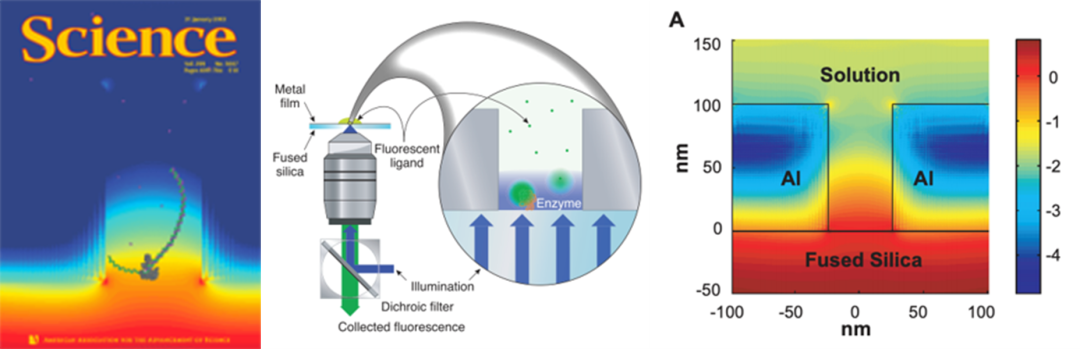
Pacific Biosciences的创始人Stephen W. Turner等科学家研究了6年之后,于2009年1月在Science杂志上发表了题为“Real-Time DNA Sequencing from Single Polymerase Molecules”的文章,将零模波导技术应用到了单分子实时测序,如下图所示,单个DNA聚合酶被固定在微孔的底部,在边合成边测序的过程中,加入不同荧光颜色标记的四种单核苷酸,当某个核苷酸形成碱基互补配对时,微孔板的底部(即限制了激发光的区域)会产生瞬时的荧光信号,磷酸二酯键的形成会释放偶联着荧光染料的产物,荧光染料渗透出微孔,荧光信号消失,这样的一个周期形成了荧光脉冲,记录下四种不同颜色的荧光脉冲可以实时的进行DNA序列的读取,这种方法直接且速度更快。由于将DNA聚合酶固定在微孔板底部,而不是将单链DNA固定在底部,测序的读长不再受近场光的场强范围限制(数十至数百纳米),对单分子测序的读长也实现了数量级的增加。

Oxford Nanopore Technologie
在美国人类基因组研究所发布突破性特殊计划的时候,除了单分子荧光,最引人注目的方法就是通过单分子的电信号来测序的纳米孔方法。荧光信号的产生与接收都需要在设备中安装复杂且体积较大的光学原件,灵敏的电学信号一直是基因测序研究者们希望用来取代光学信号的主流选择之一,遵循这个思想的还有商业化测序公司Ion Torrent,纳米孔测序技术在最开始给大家描述的场景就是一个U盘大小的装置,滴上溶液后直接插在电脑上就可以完成基因测序及信号记录。
1996年的PNAS杂志中的生物物理板块发表了一篇名为“Characterization of individual polynucleotide molecules using a membrane channel”的文章,文章中第一次记录了单个单链核酸分子在穿过纳米孔时产生的电流变化,通过电场驱动单链核酸分子通过一个直径2.6纳米的磷脂双分子层的离子通道,由于通道的直径只能允许一个单链的核酸分子通过,当单分子通过时会阻挡电流通路升高了电阻,从而产生一个电流的下降,下降延续的时间正比于通过纳米孔的单分子的长度,而这篇文章所讲的离子通道就是现在常用的α-溶血素。
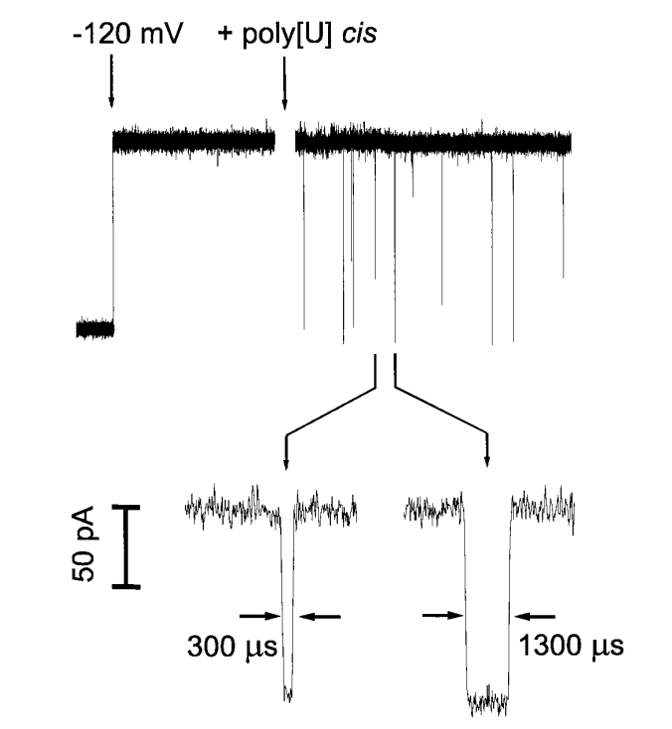
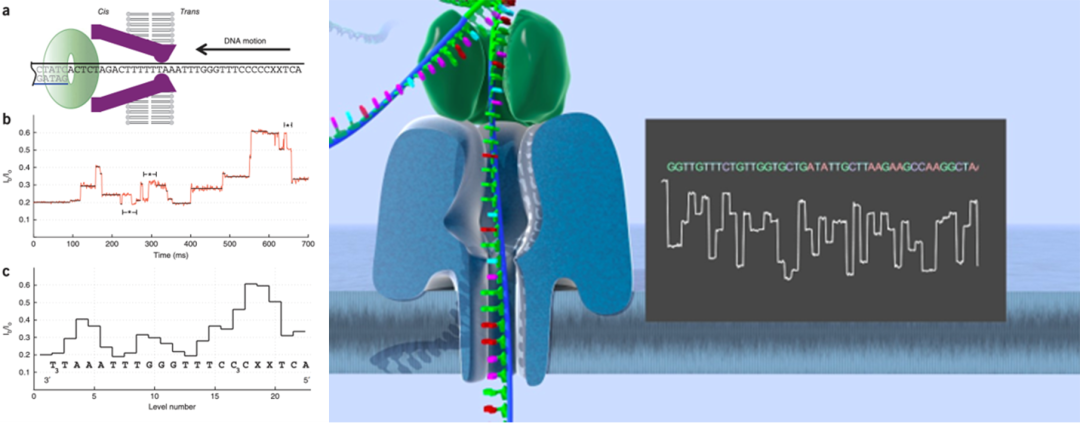
很长一段时间,纳米孔测序都徘徊在检测重复的相同碱基,因为单链核酸通过纳米孔的速度非常快,高频的弱信号很难被捕获且清晰的处理分析,直到两篇文章的发表这一技术才得到飞跃性进展,文章分别是2008年在PNAS上发表的“Single-molecule DNA detection with an engineered MspA protein nanopore”以及2012年在Nature Biotechnology上发表的“Reading DNA at single-nucleotide resolution with a mutant MspA nanopore and phi29 DNA polymerase”。MspA的工程化提供了更短且狭窄的离子通道,phi29 DNA聚合酶的加入控制了DNA通过孔的转运速度,像步进电机一样将单链核酸分子逐个碱基拉入纳米孔中,电流信号也得到了增强,第一次真正意义上实现了单核苷酸灵敏度的纳米孔测序。
文章供稿:彩科生物





 个人中心
个人中心
 我是园区
我是园区




