
2006年, 四个独立研究组同时报道了一类新小分子非编码RNA,因为它们特异地与PIWI家族蛋白相互作用,所以被命名为PIWI-interacting RNA,简称piRNA。与microRNA
(miRNA)
和small interfering RNA
(siRNA)
相比,piRNA更长,其长度在30 nt之间,并且3′末端有2′-O-甲基化修饰。从加工途径来看,miRNA和siRNA分别由RNase III
(Dicer)
切割发夹形或双链前体转录本生成;而piRNA由长单链转录本产生,且加工成熟不依赖于Dicer。
miRNA、siRNA和piRNA与Argonaute家族蛋白结合,通过碱基互补配对原则引导Argonaute蛋白靶向目的基因而发挥调控作用。真核生物Argonaute蛋白家族分为AGO亚家族和PIWI亚家族:AGO亚家族蛋白广泛表达并结合miRNA或siRNA;而PIWI亚家族蛋白特异性地在动物生殖细胞中表达并与piRNA结合。PIWI-piRNA复合物的经典功能是沉默转座元件
(transposable element,TE)
、维持生殖细胞基因组稳定性和完整性。然而,许多piRNA序列来源于非转座元件序列,这意味着piRNA的功能不仅仅是沉默转座元件。越来越多的证据表明,PIWI-piRNA复合物也参与生殖细胞中蛋白编码基因的表达调控。近年来的大量研究系统地揭示了PIWI–piRNA复合物在动物生殖细胞的生物学功能。从节肢动物到哺乳动物,piRNA通路关键因子被证明对精子发生、卵子发生或二者都至关重要,虽然在不同模式动物的作用会有所不同。此外,PIWI-piRNA功能异常也被发现与多种人类疾病密切相关,包括生殖障碍、肿瘤及神经退行性疾病等。
2022年9月14日,中国科学院分子细胞科学卓越创新中心
(生物化学与细胞生物学研究所)
刘默芳研究组和法国蒙彼利埃大学人类遗传学研究所Martine Simonelig教授团队在Nature Reviews Molecular Cell Biology在线发表了题为
Emerging roles and functional mechanisms of PIWI-interacting RNAs
的长文综述论文,
系统总结了piRNA簇转录、piRNA生成、piRNA通路机制与功能,概述了piRNA在果蝇和小鼠生殖细胞发育及人类疾病中的新作用。
大量piRNA是从称为piRNA 簇
(piRNA clusters)
的特定基因位点产生的,piRNA 簇经 RNA 聚合酶 II
(Pol II)
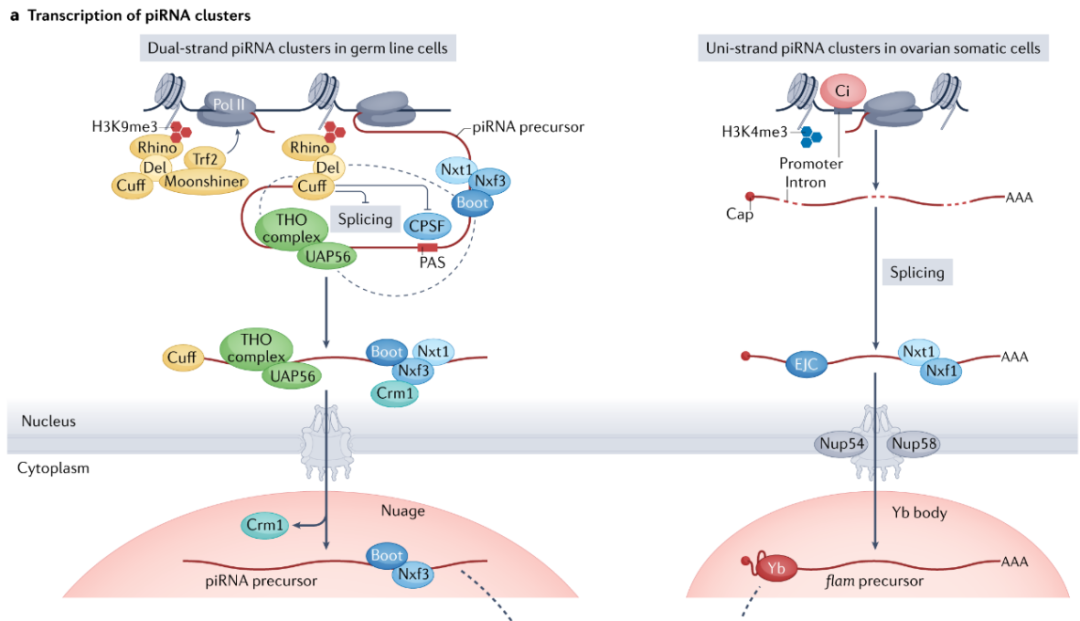
转录为长链非编码转录本。目前对 piRNA簇转录的认识主要来自果蝇中的研究,果蝇piRNA簇位于着丝粒周围和端粒基因座中,并且大部分由转座元件组成。
果蝇雌性生殖细胞中的大多数 piRNA 簇都是双向的,缺乏明确的启动子区域,并存在转录抑制相关的H3K9me3修饰。piRNA簇如何克服H3K9me3的转录抑制作用是领域的难点问题,而最近的系列性研究进展回答了这一问题
(图1)
。Rhino 是一种果蝇异染色质蛋白 HP1的同源蛋白,通过与 H3K9me3结合并募集蛋白Deadlock 和 Cutoff形成 Rhino-Deadlock-Cutoff
(RDC)
复合物
(图1a, 左)
。RDC复合物通过非经典机制激活转录,在双链 piRNA 簇的转录中起关键作用。Deadlock 招募了转录起始因子 Moonshiner,进而结合 TATA box结合蛋白相关因子2
(Trf2)
,介导Pol II转录。此外,Cutoff 结合新生成的 piRNA簇转录本并通过抑制剪接和多聚腺苷化促进转录:Cutoff通过募集转录和出核
(transcription and export,TREX)
复合物
(包括 UAP56 和 THO 复合物)
抑制 piRNA 前体剪接,并通过抑制CPSF与poly (A) 位点结合来阻断 3' 端切割和多聚腺苷化,然后将产生的 piRNA 前体转录本转运到称为nuage的核外周 RNP 颗粒。此外,核输出复合物变体Nxf3–Nxt1也参与了piRNA 前体转录本的出核转运:Nxf3 被RDC 复合物相互作用蛋白Bootlegger 募集到 piRNA 前体转录本,与输出蛋白 Crm1 结合并帮助piRNA 前体转录本出核。
与雌性生殖细胞一样,果蝇雄性生殖细胞中的 piRNA 簇也是双向的。最近的一项研究表明,RDC 复合物也参与了雄性生殖细胞中piRNA 簇的转录
【1】
。另一项研究表明,尽管不同性别果蝇含有特定的piRNAs以沉默性别特异性转座元件,但大多数 piRNA 簇在雄性和雌性果蝇之间是共享的
【2】
。
果蝇卵巢体细胞中的 piRNA 簇是单向的并且有独立的启动子
(图 1a,右)
。研究得最透彻的果蝇卵巢体细胞piRNA 簇是X 染色体上的 flamenco
(flam)
。 flam基因位点没有 H3K9me3标记,转录出来的转录本也被正常剪接。经典核输出复合物 Nxf1–Nxt1 与外显子连接复合物均介导了flam 转录本的核输出。最新研究表明,特定的核孔蛋白Nup54 和 Nup58 与Nxf1-Nxt1复合物相互作用参与了flam转录本的核输出
【3】
。
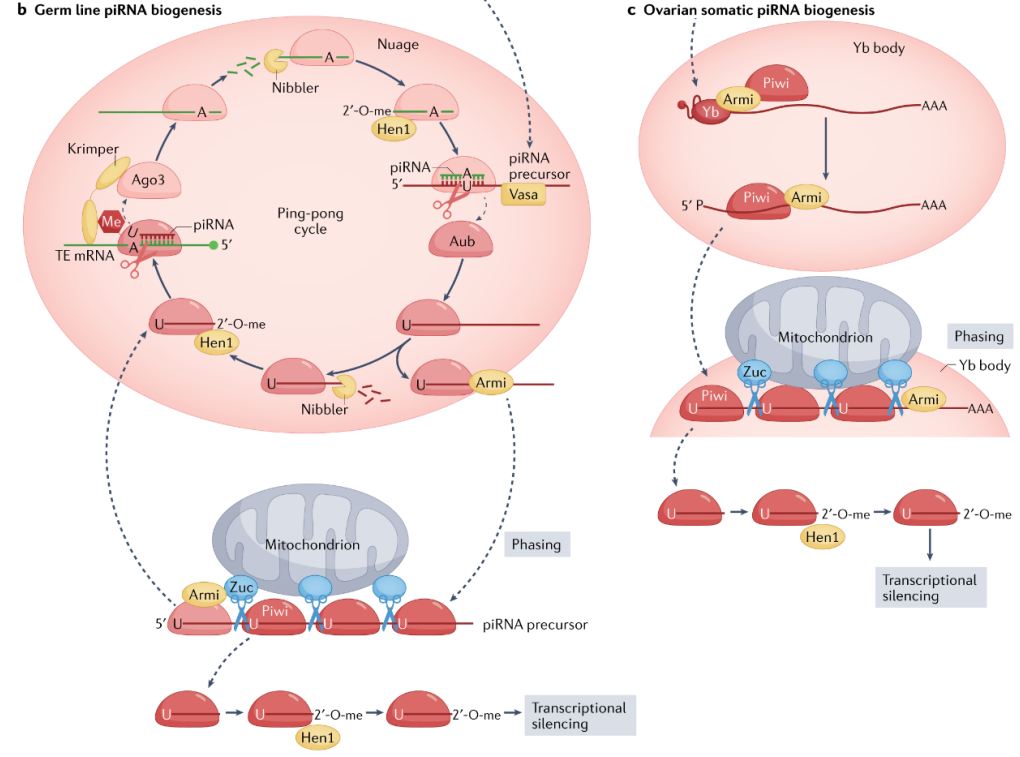
有趣的是,与果蝇同时存在单链和双链 piRNA 簇不同,小鼠缺乏 RDC 复合物和双链 piRNA 簇
(图 2)
。随着雄性生殖细胞发育和分化,小鼠表达了三种不同类型的 piRNA,分别被称为“胎儿piRNA”、“前粗线期 piRNA”和“粗线期 piRNA”。胎儿 piRNA 与 小鼠PIWI 蛋白 MILI 和 MIWI2 结合,在胎儿期睾丸中发挥沉默转座元件的功能;前粗线期 piRNA在减数分裂粗线期之前表达,与MILI相互作用;粗线期 piRNAs 主要来自基因间区域,与 MIWI 和 MILI结合,从减数分裂的粗线期精母细胞开始表达并持续到减数分裂后的延长形精子细胞中。在小鼠中,已经发现了 100 多个粗线期 piRNA 簇,它们几乎都受转录因子 A-MYB 激活并由 Pol II 转录
(图2a)
。BTBD18 是小鼠粗线期精母细胞核蛋白,促进了一部分粗线期 piRNA 簇的转录延伸。粗线期 piRNA 前体转录本包含 5' 帽子和 3' poly(A) 尾,与THO 复合物相互作用。THO复合物也参与了粗线期 piRNA 簇的转录延伸和转录本的细胞核输出。
piRNA前体转录本从细胞核转运到细胞质后,就定位到靠近细胞核的RNP颗粒,在那里它们被加工为成熟piRNA。最近的一项研究表明,piRNA生成在物种间保守,并提出了 piRNA 生成的通用模型
【4】
。该模型涉及两个相互关联的机制:发生在RNP颗粒中的Ping-pong循环途径和发生在线粒体外膜的Phasing加工途径。
在果蝇中,Ping-pong循环依赖于两种胞质定位 PIWI 蛋白Aub 和 Ago3之间的相互协作
(图1b)
。Ping-pong循环由母源获得的Aub-piRNA启动,由其识别互补的 piRNA 前体,并将它们在对应于向导piRNA 5' 端第10位和第11位核苷酸位点切割,产生次级piRNA单磷酸化的 5' 端;然后,Ago3 结合到次级piRNA 5' 端,该RNA片段下游再被Aub、Ago3 或者Zucchini进行切割,产生 3' 端;随后,3' 到 5' 核酸外切酶 Nibbler对3' 端剪切;最后,3' 末端被甲基转移酶 Hen1
(小鼠中为 HENMT1)
进行2'-O-甲基化修饰,产生成熟的 piRNA
(图1b和2c)
。在接下来的Ping-pong循环中,装载piRNA 的Ago3 靶向并切割互补的 piRNA 前体,生成一个新的单磷酸化5' 末端,该末端与 Aub 结合;然后,该 piRNA 前体下游被切割,并在进入新循环之前完成3' 端剪切和2'-O-甲基化修饰
(图1b)
。细胞质 PIWI 蛋白的相互协作导致产生具有 10 个核苷酸重叠的互补 piRNA,装载到 Aub 的 piRNA 具有 1U
(尿嘧啶)
偏好性,而装载到Ago3 的 piRNA 具有 10A
(腺嘌呤)
偏好性。
piRNA Phasing加工涉及 nuage 和线粒体定位因子间的相互作用,并导致沿着piRNA 前体产生一系列头尾相接的phased piRNA
(图 1b 和 2c)
。Aub与 5' -单磷酸化piRNA 前体结合后,在 nuage 中与 RNA 解旋酶 Armitage(Armi;小鼠中为 MOV10L1)相互作用;然后,Armi将复合物转移到线粒体外膜,其中Zucchini(小鼠中为PLD6)切割piRNA前体产生前一个piRNA的 3' 末端和下一个piRNA的5'末端。之后,piRNA 前体剩余部分在其 5' 末端被 Piwi(小鼠中为 MIWI2)反复结合,并被Zucchini反复切割,产生一串头尾相接的 piRNA
(图 1b 和 2c)
。Zucchini具有U前切割偏好性,且 Piwi 偏好结合以 U 开头的 RNA 片段,这导致在phased piRNA中产生了强烈的 1U 偏好性。Phasing加工途径通过产生新piRNA 来增加 piRNA多样性,而Ping-pong循环途径则增加已存在piRNA的丰度。
piRNA 前体转录本如何被选择进入piRNA加工途径长期未知。最近的研究表明,极长的第一个外显子和特定的表观遗传标记是哺乳动物 piRNA 簇的保守特征
【5】(图2a)
。此外,出乎意料的是,核糖体也被报道参与粗线期piRNA生成
(图 2b)
,并且发现核糖体印迹可以决定 piRNA 加工位点。这种机制在小鼠和鸡中保守,提示piRNA 生成机制和翻译之间存在紧密联系。
与miRNA类似,piRNA的靶向作用
(切割或仅结合不切割)
取决于其与靶RNA的碱基互补配对程度。PIWI-piRNA复合物最早报道可直接切割靶RNA,类似于siRNA。通过生信和报告基因分析,piRNA介导的切割规则已逐渐清晰。高效靶向切割需要piRNA第2-11位碱基与靶RNA完全匹配、第12-22位碱基的附加配对
(允许一些错配)
,而piRNA的第1位碱基和3′区域的互补配对为靶向切割非必需。
在秀丽隐杆线虫中,piRNA以miRNA类似的碱基互补配对原则靶向生殖细胞mRNA:第2-8位碱基
(Seed区域)
的完美配对与第14-19位碱基的附加配对。然而,由于秀丽隐杆线虫piRNA
(21 nt)
较短,其他物种中较长的piRNA可能具有不同的互补配对规则。在果蝇卵巢中,Aub结合的piRNA利用较短的Seed互补配对序列
(2-6 nt)
和较长的附加配对序列
(16-24 nt)
来识别靶mRNA。这种Aub-piRNA靶向的mRNA可被捕获到卵母细胞后极的胚质中,而这种靶向模式不会导致mRNA切割。但是,Aub–piRNA靶向那些位于胚胎胚质外的相同mRNA,则会导致它们通过脱腺苷化降解,显示piRNA引导的PIWI蛋白可招募各种不同效应因子到mRNA发挥不同效应。同样,在小鼠延长型精子细胞中,通过piRNA与靶mRNA不完全互补配对,MIWI/piRNA可靶向识别大量mRNA,这种碱基互补配对不会导致靶mRNA直接的切割降解,而是通过招募脱腺苷酶CAF1使靶mRNA发生脱腺苷化降解。尽管这些研究发现了piRNA会因为与靶基因的碱基配对程度不同而产生不同的作用结果,但piRNA的精确靶向规则目前仍然不清楚。
沉默转座元件。PIWI-piRNA复合物最初发现的功能是沉默转座元件、维持动物生殖细胞基因组稳定性和完整性。PIWI蛋白与piRNA 形成的不同复合物可分别在转录水平和转录后水平沉默转座元件。细胞质 PIWI 蛋白,如果蝇Aub和Ago3、小鼠 MIWI 和 MILI,主要在转录后水平经piRNA靶向切割转座元件转录本。而核定位PIWI 蛋白,包括果蝇 Piwi 和小鼠 MIWI2,在装载了Phased piRNA后得以入核,通过向导piRNA与转座元件新生转录本互补配对,识别转座元件位点,并募集相关因子对转座元件进行转录水平沉默
(图 3)
。在果蝇中,通过遗传筛选已经鉴定出一系列参与转录沉默的 Piwi 辅助因子。在果蝇卵巢体细胞中,Maelstrom
(Mael)
通过与 SWI-SNF 染色质重塑复合物相互作用,减少染色质松弛程度从而抑制转座元件转录
(图 3a)
。Panoramix
(Panx;也称为Silencio)
在转座元件沉默中具有核心作用。Panx与Nxf2和Nxt1形成三元复合物,Nxf2结合新生 RNA 并帮助Piwi锚定到新生 RNA上,而 Panx 则招募异染色质重塑因子来沉默转座元件。进一步的研究证明,动力蛋白轻链 LC8
(Cut up)
可以介导Panx-Nxf2-Nxt1复合物二聚化
(图 3a)
。Panx依赖性异染色质形成需要 H3K9 甲基转移酶 Eggless
(Egg;也称为 SetDB1)
及其辅助因子 Windei
(Wde)
,以及 H3K4 去甲基化酶 Lsd1。核小体重塑和去乙酰化酶
(NuRD)
复合物成分 Mi-2 和 Rdp3也参与了 Piwi 依赖性转录沉默。最后,SUMO 连接酶 Su(var)2-10 通过将 Piwi 复合物
(包括 Arx和 Panx)
与异染色质机制连接,在转座元件转录抑制中发挥关键作用
(图 3a)
。
在小鼠中,除了 H3K9me3外,MIWI2-piRNA 依赖性转录沉默还涉及 DNA 甲基化
(图 3b)
。piRNA 介导的DNA甲基化发生在胎儿期生殖母细胞中。最近的一项研究表明,MIWI2相互作用蛋白 SPOCD1 为生殖母细胞中转座元件甲基化所必需
【6】
。SPOCD1 与 DNA 甲基转移酶、染色质重塑蛋白 NuRD和 BAF相互作用,共同在转录水平沉默转座元件
(图 3b)
。最近还有报道,piRNA 复合物的另一个辅助因子 TEX15,通过与MIWI2
【7】
或MIL
I
【8】
的相互作用,介导piRNA 依赖的转座元件DNA甲基化
(图 3b)
。
图3:PIWI-piRNA复合物转录水平沉默转座元件的分子机制
调控蛋白编码基因。除了沉默转座元件外,piRNA 通路还通过不同的分子机制在生殖发育过程中调控蛋白编码基因
(图 4)
。该功能首次在果蝇胚胎中被报道,其中转座元件来源的piRNA被证明可将 Aub引导至nanos mRNA,随后Aub通过招募 RNA 结合蛋白 Smaug和CCR4-NOT脱腺苷化复合物,介导胚胎体细胞中nanos mRNA脱腺苷化降解
(图 4a)
。在小鼠中,粗线期 piRNA与MIWI结合,通过招募脱腺苷酶 CAF1,促进了后期精子细胞中大量mRNA的脱腺苷化降解
(图 4b)
。此外,粗线期 piRNA 还可以指导 MIWI 通过类似 siRNA 的机制直接切割靶mRNA
(图 4b)
。同样的,在果蝇早期胚胎中,装载piRNA的Aub也可以直接切割降解体细胞中的母源mRNA
(图 4a)
。
有趣的是,最近的一项研究表明,MIWI 和粗线期 piRNA 通过不完全互补配对的方式,激活了一组球形精子细胞mRNA的翻译,该机制需要MIWI与翻译起始因子eIF3F和RNA结合蛋白 HuR形成翻译激活超级复合物MIWI-eIF3F-HuR
(图 4b)
。随后,在果蝇胚胎中也发现了类似的机制,其中 Aub 与poly(A)结合蛋白PABP和几个 eIF3 亚基相互作用,激活胚胎生殖细胞nanos mRNA的翻译起始
(图 4a)
,显示了PIWI-piRNA复合物在激活翻译中的进化保守性。
领域内的这些发现扩展了piRNA调控蛋白质编码基因的多样性,但这些调控背后的机制仍不完全清楚。对编码基因的调控方式取决于靶mRNA与piRNA的碱基互补配对水平,比如切割需要更高的互补性。然而,在非切割情况下,PIWI 蛋白需要招募不同的辅助因子实现对mRNA 的不同调控作用。
图4:PIWI-piRNA复合物调控蛋白编码基因的分子机制
大量动物遗传学模型研究表明,piRNA调控通路为动物生殖发育必需,其中piRNA调控通路对斑马鱼、果蝇及仓鼠的雌、雄性生殖发育均必需,而在小鼠中,piRNA调控通路仅为雄性生殖发育必需
(如表1所示)
。
早期对果蝇遗传学的研究发现,三种PIWI蛋白在果蝇原始生殖细胞的形成和发育、生殖干细胞的自我更新和分化、以及卵母细胞轴的确定中发挥作用,缺失任一种PIWI蛋白均会导致雌性和雄性果蝇生殖发育异常。在小鼠中,PIWI蛋白和piRNA在睾丸中高表达,而在卵巢中无明显表达;与之一致的的是,Miwi、Mili、Miwi2或其他关键piRNA通路基因的缺失致雄性不育,但不影响雌性生育。
虽然piRNA调控通路的基因敲除动物模型有力地支持了piRNA在动物生殖发育中的重要作用,但piRNA簇的缺失可为piRNA的生物学功能提供更直接的证据。在果蝇中,piRNA簇Su(Ste)和flam的缺失分别导致雄性和雌性不育,这支持了不同piRNA发挥不同作用的观点。同样的,尽管缺失piR2或piR17 piRNA簇的小鼠表现出正常的生育能力,但缺失piR6 piRNA簇的雄鼠生育力低下,缺失piR18 piRNA簇的雄鼠不育。
近期,三个独立的研究组报道了PIWI蛋白和piRNA在仓鼠卵母细胞发育中发挥重要作用
(如表1所示)
。之前有研究报道,在不同哺乳动物的雌性生殖细胞中均存在piRNA,且发现哺乳动物雌性生殖细胞中存在一种特有的PIWIL3-piRNA复合物
【9,10】
。实际上,大多数哺乳动物基因组编码4个PIWI基因(PIWIL1-PIWIL4),而小鼠和大鼠基因组中缺乏PIWIL3。在仓鼠中,PIWIL3只在MII卵母细胞中结合piRNA,同时PIWIL3高度磷酸化。有趣的是,PIWIL3结合的piRNA与精子细胞中的piRNA不同,其长度为19 nt,具有1U偏好性,但其3′端缺乏2'-O-甲基化修饰。此外,PIWIL1在仓鼠睾丸和卵巢中均表达,并在MII卵母细胞中结合了两种不同长度的piRNA
(29-30和22-23 nt)
。更重要的是,在仓鼠中敲除Piwil1或Mov10l1会导致雄性和雌性完全不育,而敲除Piwil3则会降低雌性的生育能力而不影响雄性。这些研究进一步证明,PIWI-piRNA复合物在哺乳动物两性生殖中均发挥功能,对我们理解人类piRNA作用具有重要意义。
piRNA调控异常与男性不育。鉴于piRNA在动物生殖生育过程中的重要作用,近年来许多研究者在鉴定不育症患者中PIWI通路基因突变方面做了大量的工作。最初在男性不育患者中发现了PIWIL1、PIWIL2、PIWIL3和PIWIL4的单核苷酸多态性。更为重要的是,最近一项研究在无精症患者中发现的PIWIL1泛素化降解缺陷的基因突变,利用基因突变敲入小鼠模型证明,此类突变致雄性不育,证明这类突变是男性不育致病性突变;机制研究发现,同源突变导致MIWI在小鼠后期精子细胞中异常积累,抑制了组蛋白泛素连接酶RNF8的入核,进而抑制了精子细胞组蛋白-鱼精蛋白的交换。这些发现证实了PIWI基因突变为男性不育致病突变。
近期一项研究报道了4例无血缘关系的中东裔不育男性携带了piRNA加工酶基因PNLDC1突变
【11】
。另一项相关研究也在一名中国汉族不育男性中发现了纯合PNLDC1 R47G突变,该突变显著损伤了PNLDC1加工piRNA的活性,并在小鼠模型证明此突变与人类男性不育的因果关系
【12】
。
拟染色质小体
(Chromatoid body,CB)
是球形精子细胞中的一种典型无膜结构,也是piRNA复合物富集及功能场所。TDRD7为CB结构所必需,也是piRNA调控通路关键功能蛋白。有研究在男性不育症患者中发现了TDRD7的纯合无义突变,TDRD7敲入突变小鼠再现了患者的疾病表型,提示这些为男性不育致病突变。此外,对近亲家系分析发现了不育症患者携带的多种piRNA通路基因有害突变,包括纯合TEX15无义突变、TEX15复合杂合突变、TDRD6纯合错义突变和TDRD9纯合移码突变等。尽管已在不育男性中发现了大量的piRNA通路基因的遗传突变,但目前只有少量基因突变在动物模型中被证明会导致不育,其因果关系仍有待确定。
piRNA调控异常与其他人类疾病。大量研究发现,PIWI基因在多种人类肿瘤中被异常激活。尽管越来越多的证据表明PIWI蛋白在癌症发生发展中发挥促癌作用,然而它们在癌细胞中是否会与piRNA一起发挥功能仍有争议。也有很多报道在多种肿瘤组织和细胞系鉴定到了类似piRNA序列的小RNA,然而并没有充足的证据证明这些小RNA是否是真实的piRNA分子?
有趣的是,两个独立研究组近期报道了在人类癌症中异常表达的PIWIL1以piRNA非依赖的方式调控肿瘤发生发展。在胰腺癌细胞中,PIWIL1作为APC/C泛素连接酶的一个共激活因子,触发了细胞黏附蛋白Pinin的泛素化降解,从而促进胰腺癌转移。同样,PIWIL1在胃癌中以piRNA非依赖的方式与包括UPF1在内的无义mRNA降解因子相互作用,介导靶mRNA的降解。相比之下,敲除结肠癌细胞系中异常表达的PIWIL1并不影响转座元件的表达和癌症的发生发展,这表明PIWIL1的促癌作用仅限于特定的肿瘤或癌细胞系。PIWI蛋白在特定癌症中的功能是否依赖于piRNA还需要进一步研究。
除了在癌细胞中异常表达外,PIWI蛋白和piRNA还在其他组织表达,比如神经系统。海兔的神经系统有低丰度的piRNA表达,并且这些piRNA仍具有1U偏好性、3′端2'-O-甲基化修饰和特定长度。更为重要的是,有研究报道神经细胞中PIWI蛋白和piRNA为神经元发育、学习和记忆必需。此外,piRNA通路基因突变与人类神经退行性疾病也有一定相关性。
虽然PIWI蛋白和piRNA在人类疾病中的潜在作用已引发了研究人员的极大兴趣,但是到目前为止,大多数研究还仅是相关性研究。未来在模式生物中的遗传学研究将有助于明确PIWI-piRNA在动物生殖系以外的生物学功能及作用机制。
近年来,piRNA领域研究已取得了诸多突破性进展,有效地拓展了对piRNA簇的转录、piRNA生成、piRNA作用机制和生物学功能的理解。但piRNA领域仍有许多未解之谜。例如,piRNA的部分生成发生在线粒体外膜上,但piRNA生成与线粒体之间的关系尚不清楚,piRNA生成是否需要线粒体提供能量?此外,piRNA和核糖体之间的复杂关系还了解不多,核糖核蛋白复合物如何参与piRNA的生成和功能?反过来,piRNA前体和成熟piRNA如何影响核糖核蛋白复合物的生物特性?由于适应性进化,piRNA显示出高度的不保守性,甚至在同一个物种的不同群体间也如此,但依赖于piRNA的mRNA调控过程在亲缘关系很远的物种间却很保守,如何解释这一矛盾?另外,PIWI-piRNA生殖系外的体细胞功能及PIWI蛋白的piRNA非依赖性功能等,都将成为领域未来研究的新方向和新增长点。
原文链接:
https://www.nature.com/articles/s41580-022-00528-0
1. Chen, P., Luo, Y. & Aravin, A.A. RDC complex executes a dynamic piRNA program during Drosophila spermatogenesis to safeguard male fertility. PLoS Genet 17, e1009591 (2021).
2. Chen, P. et al. piRNA-mediated gene regulation and adaptation to sex-specific transposon expression in D. melanogaster male germline. Genes Dev 35, 914-935 (2021).
3. Munafo, M. et al. Channel nuclear pore complex subunits are required for transposon silencing in Drosophila. eLife 10 (2021).
4. Gainetdinov, I., Colpan, C., Arif, A., Cecchini, K. & Zamore, P.D. A Single Mechanism of Biogenesis, Initiated and Directed by PIWI Proteins, Explains piRNA Production in Most Animals. Mol Cell 71, 775-790 e775 (2018).
5. Yu, T. et al. Long first exons and epigenetic marks distinguish conserved pachytene piRNA clusters from other mammalian genes. Nat Commun 12, 73 (2021).
6. Zoch, A. et al. SPOCD1 is an essential executor of piRNA-directed de novo DNA methylation. Nature 584, 635-639 (2020).
7. Schopp, T. et al. TEX15 is an essential executor of MIWI2-directed transposon DNA methylation and silencing. Nat Commun 11, 3739 (2020).
8. Yang, F. et al. TEX15 associates with MILI and silences transposable elements in male germ cells. Genes Dev 34, 745-750 (2020).
9. Roovers, E.F. et al. Piwi proteins and piRNAs in mammalian oocytes and early embryos. Cell Rep 10, 2069-2082 (2015).
10. Williams, Z. et al. Discovery and Characterization of piRNAs in the Human Fetal Ovary. Cell Rep 13, 854-863 (2015).
11. Nagirnaja, L. et al. Variant PNLDC1, Defective piRNA Processing, and Azoospermia. N Engl J Med 385, 707-719 (2021).
12. Wang, X., Tan, Y.Q. & Liu, M.F. Defective piRNA Processing and Azoospermia. N Engl J Med 386, 1674-1675 (2022).
【非原创文章】本文著作权归文章作者所有,欢迎个人转发分享,未经作者的允许禁止转载,作者拥有所有法定权利,违者必究。










 药选址
药选址







