
癌症是由渐进的基因和表观遗传变化驱动,在整个过程中,癌细胞可以获得复杂的异质性,进而更具侵袭性和转移性,并扩散到身体其他部位形成新的肿瘤,加速疾病的进程。因此,深入了解肿瘤亚克隆选择和转移的分子基础、转录状态的起源和转变以及肿瘤进化路径的遗传决定因素,不仅有助于阐明肿瘤进化的基本原则,还具有临床意义。
基因工程小鼠模型(Genetically engineered mouse models, GEMMs)是研究肿瘤进展的一个关键工具,研究人员能够通过GEMMs研究肿瘤在原生微环境和实验定义的条件下的演化过程。其中,Kras
LSL-G12D/+
; Trp53
fl/fl
(KP)模型通过病毒传递Cre重组酶到少量肺上皮细胞引发肿瘤,导致致癌基因Kras的激活、P53肿瘤抑制基因的纯合缺失和肿瘤的克隆生长等,真实模拟了新生细胞转化成侵袭性转移肿瘤的主要步骤,从分子和组织病理学上再现了人肺腺癌的进展。因此,我们可以通过KP模型来探究肿瘤演变过程中尚未解决但非常关键的问题。
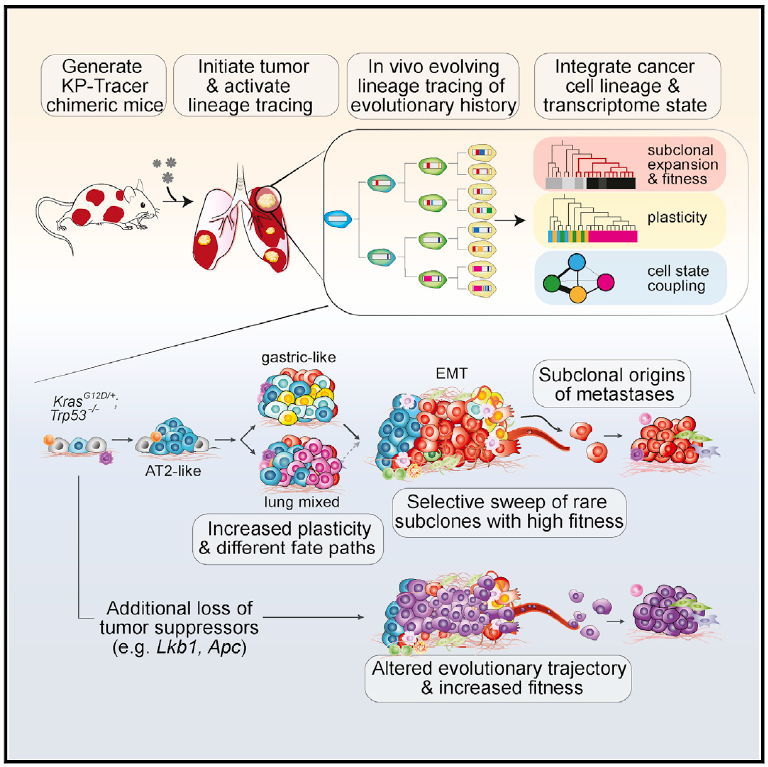
近日,美国加州大学Jonathan S. Weissman研究团队及合作者在Cell上发表了题为“Lineage tracing reveals the phylodynamics, plasticity, and paths of tumor evolution”的文章。研究团队将基于单细胞RNA-seq的进化谱系示踪系统引入KP小鼠模型中,连续并全面监测了一个携带致癌突变的单细胞演变为侵袭性肿瘤的全过程,揭示罕见的亚克隆可以通过独特的转录程序驱动肿瘤扩张。此外,研究团队还发现肿瘤通过典型、独特的进化轨迹发展,干扰额外的肿瘤抑制因子可以加速肿瘤的进展。该研究以前所未有的规模和分辨率重建了从单一转化细胞到复杂、侵袭性肿瘤群体的肿瘤演化全过程。
KP-Tracer小鼠可以连续和高分辨率追踪肿瘤的起始和进展
为生成高分辨率的肿瘤演化系统,研究团队开发了一种具有谱系追踪能力的肺腺癌小鼠模型KP-Tracer,能够连续数月进行细胞谱系追踪。后续实验证实,在5-6个月后,该模型成功追踪了肿瘤发生,并且示踪剂能够在相应部位表达。此外,在对癌细胞进行单细胞转录组测序分析后,发现细胞状态、谱系、样本身份和肿瘤克隆性在肿瘤中的表达与预期一致。
图1. KP-Tracer小鼠模型的构建。来源:Cell
肿瘤进化中的一个关键问题是,基于肿瘤生长促进基因或表观遗传变化的亚克隆选择以及由此产生的亚群动态变化如何导致侵略性亚克隆对同一肿瘤的其他部分的扩展。为研究KP肿瘤的亚克隆动力学,研究团队采用了一种统计检验方法,即将每个亚克隆的相对大小与没有亚克隆被选择的“中性”进化模型中的大小进行比较分析。结果显示,有些肿瘤似乎是中性进化的,即没有证据表明阳性选择;有些亚克隆则显示出明显的阳性选择迹象。
此外,研究团队发现肿瘤主要由一个(有时两个)正在扩增的亚克隆驱动。在肿瘤中,扩增细胞的比例分布广泛, 表明了亚克隆扩展的侵袭性;扩增细胞以增加的DNA拷贝数变异、细胞周期评分和适应度评分为标志。
图2. 罕见亚克隆的显著扩增及其特性。来源:Cell
绘制细胞状态之间的系统发育关系揭示肿瘤进化的共同路径
原则上,KP模型中观察到的细胞可塑性、转录异质性可能来自于通过转录状态的随机或结构化进化路径。为了研究肿瘤进化路径的一致性,研究团队开发了一个称为“进化耦合”的统计数据,扩展了克隆耦合统计数据来量化成对细胞状态之间的系统发育距离。基于不同转录状态的占比和进化耦合的全套肿瘤的数据驱动分层聚类显示,肿瘤可以分为三个不同的组(Fate Cluster1、Fate Cluster2及Fate Cluster3)。
Fate Cluster1、2之间共享一些转录状态,Fate Cluster1主要通过包括胃样和内胚层样状态进化;Fate Cluster2通过肺混合状态进化,Fate Cluster3以高适应度状态为主,如前上皮间质转化(Pre-EMT)和间质状态
。
进一步,研究团队开发了“Phylotime”对Fate Cluster 1、2背后的转录变化进行分析。分析结果证实,Fate Cluster 1、Fate Cluster2是两条独立的进化途径,并且每条途径显示出与Phylotime相关的不同转录变化。上述结果表明,KP肿瘤可能主要通过两种途径进化,一条是胃样和内胚层样状态,另一条是肺混合状态,且每种进化轨迹都显示出明显的转录变化。
图3. 细胞状态之间系统发育关系的构建。来源:Cell
肿瘤抑制因子的缺失会改变肿瘤的转录组、可塑性和进化轨迹
肿瘤抑制基因可以调节多种细胞活动,其丧失与肿瘤侵袭性的增加有关,但这些基因对体内肿瘤进化动力学的影响目前尚不清楚。因此,研究团队结合基因干预和定量系统动力学方法探索了额外的致癌突变如何改变KP肿瘤的进化轨迹,重点研究了人类肺腺癌中两种频繁突变的肿瘤抑制因子LKB1和APC,以及经CRISPR sgRNA敲除LKB1和APC后产生两种动物模型(KPL和KPA)。结果显示,靶向LKB1或APC会增加肿瘤负担,但亚克隆扩增的数量和相对大小没有改变;与肿瘤适应性相关的基因在遗传背景中差异较大。
图4. 遗传扰动会改变肿瘤的转录适应性和可塑性。来源:Cell
为检测LKB1和APC的异常是否改变了KP肿瘤的转录图谱,研究团队整合了KPL、KPA肿瘤和之前的KP肿瘤的单细胞转录组数据集。结果显示,经额外的LKB1和APC干扰后产生了四个新的转录状态。此外,针对LKB1/APC的干预也导致主导转录组状态的改变:KPL肿瘤主要富集在上皮细胞-间充质转化前状态(Pre-EMT),KPA肿瘤富集在APC特异性早期、间质和转移状态。
为研究肿瘤抑制因子的缺失如何改变进化轨迹,研究团队对单个肿瘤的转录状态占比和进化耦合进行了主成分分析。结果显示,靶向性肿瘤抑制因子LKB1或APC均可促进肿瘤生长,但其对细胞状态、可塑性和进化路径的影响差异较大 。具体而言,KPL肿瘤能够迅速发展到Pre-EMT状态下并稳定下来;KPA肿瘤则通过新的APC特异性状态开辟了一条独特的进化路径。
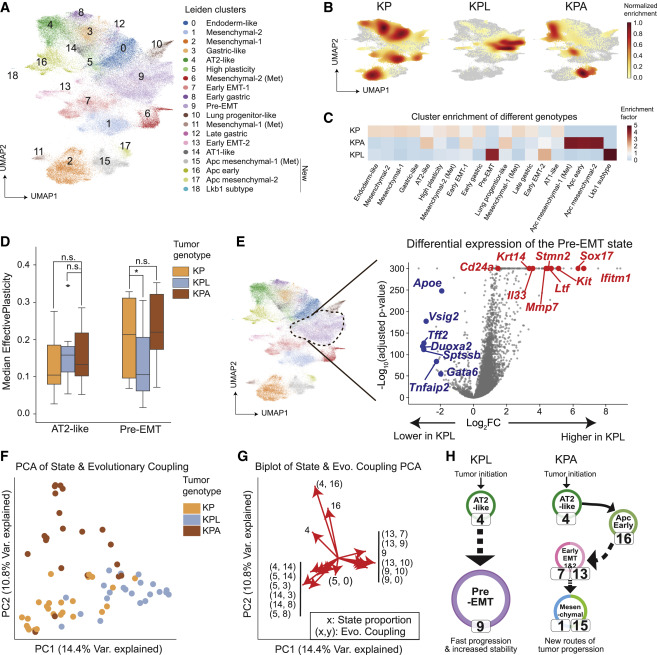
图5. 肿瘤抑制因子的缺失对肿瘤进展及细胞状态的影响。来源:Cell
综上所述,该研究首次在基因工程肺腺癌小鼠模型中使用基于CRISPR的谱系示踪剂追踪肿瘤从单一转化细胞到侵袭性肿瘤的演化过程,以连续、高分辨率的肿瘤谱系追踪为肿瘤进化建模提供了一个重要参考,绘制了从激活单个细胞的致癌突变发展成为具有侵袭性的转移肿瘤的路径图,揭示了细胞转录图谱、细胞可塑性、进化路径以及肿瘤抑制因子在肿瘤发展中的作用。
研究团队表示,随着谱系示踪工具的发展和其他新兴数据的集成,也期望该研究提出的实验和计算框架为未来构建肿瘤演化的高维、定量和预测模型奠定良好的基础,从而为新的治疗策略提供新思路。
1. Yang et al., Lineage tracing reveals the phylodynamics, plasticity, and paths of tumor evolution, Cell (2022).
2. DeTomaso, D., and Yosef, N. (2021). Hotspot identifies informative gene modules across modalities of single-cell genomics. Cell Syst.
3. Gao, R., et al. (2021). Delineating copy number and clonal substructure in human tumors from single-cell transcriptomes. Nat. Biotechnol.
目前已有500+行业精英加入基因俱乐部

























![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()




 个人中心
个人中心
 我是园区
我是园区




