
高等真核生物基因组存在复杂的三维空间结构,在不同尺度下形成染色质环(Chromatin loops)、拓扑关联结构域(TADs)、活性/非活性染色质区室(A/B compartments)和染色体域(Chromosome territories)。这些结构对于基因组稳定性的维持、基因表达的精准调控具有重要作用,从而影响细胞命运决定和表型建立。经典3D基因组结构主要通过染色体构象捕获(3C)及其衍生方法(如4Cs、5C、Hi-C)以及ChIA-PET为代表的多种形式的高通量技术揭示。这些技术可以捕获细胞核内空间相邻的成对DNA序列,但无法捕获细胞群体中基因组内协同的多位点相互作用(multi-way contact)和单分子拓扑结构(single-allele topology)。此外,基因组3D结构在细胞周期、发育和分化过程中动态变化,并与多个基因及调控区间的染色质相互作用相关。为了充分理解基因组的动态折叠机制和功能相关性,获得细胞群体中的染色体单分子拓扑结构至关重要。
近年来,多种方法如ChIA-drop、split-pool recognition of interactions by tag extension (SPRITE)、Tri-C、multi-contact 4C和Pore-C等已被建立,用于研究染色质多位点协同相互作用和群体细胞的染色体单分子拓扑结构的捕获。这些方法中,Pore-C具有技术简单、可以同步捕获全基因组高阶多位点互作信息和DNA甲基化修饰的优点。
2023年3月6日, 中山大学中山眼科中心肖传乐团队与中国科学院昆明动物研究所侯春晖团队在Nature Communications在线发表了题为“High-throughput Pore-C reveals the single-allele topology and cell type-specificity of 3D genome folding”的研究论文, 该研究优化建立了一种高通量的Pore-C方法,显著增加了高阶染色质互作的检测通量,并揭示了三维基因组的单分子拓扑结构多样性和细胞特异性。


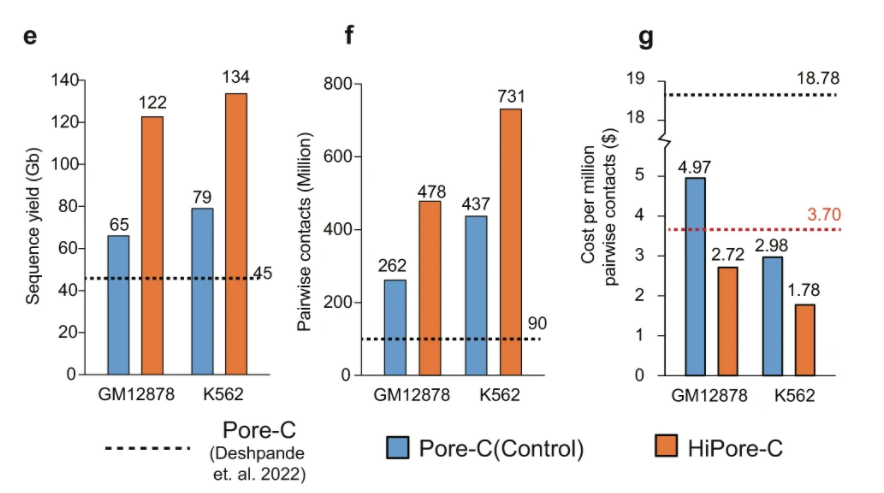
图2. HiPore-C与Pore-C技术测序通量和成本的比较。

图3. 跨越染色质环的高阶互作。

图4. K562和GM12878细胞TAD结构域的高阶互作聚类分析。

图5. K562和GM12878细胞中β-globin locus HiPore-C高阶互作分析。
中山大学中山眼科中心副研究员肖传乐和中国科学院昆明动物研究所侯春晖研究员为本文共同通讯作者,中山大学中山眼科中心博士后钟嘉泳、南方科技大学公共卫生中心牛龙见博士(中国科学院昆明动物研究所访问学者)为共同第一作者,中山大学医学院研究生林卓缤、白鑫、中山眼科中心陈颖博士、美国克莱姆森大学计算机学院罗峰教授等为该研究做出了重要贡献。
钟嘉泳博士
中山大学中山眼科中心博士后,从事三代测序数据分析工作,以第一或共一在Nature Communications、Nature Metabolism、Plos Genetics等杂志发表多篇文章。
南方科技大学公共卫生中心助理研究员,中国科学院昆明动物研究所访问学者,长期从事三维基因组学及基因表达调控的相关研究,以第一或共一在Nature Genetics、Nature Communications、Cell Reports、Communications biology等杂志发表多篇文章。
中国科学院昆明动物研究所研究员,博士生导师,中科院百人计划和国家海外高层次青年人才,专注于顺式调控元件、基因组三维结构在发育和进化过程中的动态变化及对基因的时空特异性调控机制的研究,在Nature Genetics、 Molecular Cell、PNAS、Cell Reports等杂志发表论文30余篇。
中山大学中山眼科中心副研究员,硕博士生导师,广东省杰出青年基金获得者,长期从事三代测序前沿技术开发及应用研究。主持开发了三代测序组装方法MECAT,MECAT2,NECAT和三代测序表观修饰检测方法DeepMod,在Nature Methods、Molecular Cell和Nature Communications等杂志上发表SCI论文40余篇。
【招生广告】我们实验室为生物实验和生物信息学计算提供优越的条件,同时科研经费充足(累计1000余万)。欢迎对分子医学(中山眼科中心)和计算机科学与技术(人工智能学院)方向感兴趣的生命科学、计算机科学和数学专业学生报考我们的硕博士研究生。此外,我们还欢迎计算机专业和生物信息学专业的博士毕业生到我们的实验室做博士后,我们的实验室计算和生物实验结合,博士后的产出丰硕。有意者请联系我邮箱:xiaochuanle@126.com。
目前已有500+行业精英加入基因俱乐部
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()




 个人中心
个人中心
 我是园区
我是园区









