1.Fischbach, M. A. & Walsh, C. T.Assembly-line enzymology for polyketide and nonribosomal Peptide antibiotics:logic, machinery, and mechanisms. ChemicalReviews 106, 3468-3496 (2006).
2.Gotze, S. & Stallforth, P.Structure, properties, and biological functions of nonribosomal lipopeptidesfrom pseudomonads. Nat Prod Rep 37, 29-54, doi:10.1039/c9np00022d(2020).
3.Liu, Y., Ding, S., Shen, J. &Zhu, K. Nonribosomal antibacterial peptides that target multidrug-resistantbacteria. Nat Prod Rep 36, 573-592, doi:10.1039/c8np00031j(2019).
4 .Chooi, Y. H. & Tang, Y. Addingthe lipo to lipopeptides: do more with less. Chem Biol 17, 791-793,doi:10.1016/j.chembiol.2010.08.001 (2010).
5.Baltz, R. H., Miao, V. &Wrigley, S. K. Natural products to drugs: daptomycin and related lipopeptideantibiotics. Nat Prod Rep 22, 717-741, doi:10.1039/b416648p(2005).
6.Bozhuyuk, K. A. J. et al. Modification and de novo designof non-ribosomal peptide synthetases using specific assembly points withincondensation domains. Nature chemistry11, 653-661,doi:10.1038/s41557-019-0276-z (2019).
7.Bloudoff, K. & Schmeing, T. M.Structural and functional aspects of the nonribosomal peptide synthetasecondensation domain superfamily: discovery, dissection and diversity. Biochimica et biophysica acta. Proteins andproteomics 1865, 1587-1604,doi:10.1016/j.bbapap.2017.05.010 (2017).
8.Chen, H. et al. Identification of Holrhizins E-Q Reveals the Diversity ofNonribosomal Lipopeptides in Paraburkholderia rhizoxinica. J Nat Prod 83, 537-541,doi:10.1021/acs.jnatprod.9b00927 (2020).
9 .Wang, X. et al. Discovery of recombinases enables genome mining of crypticbiosynthetic gene clusters in Burkholderiales species. Proc Natl Acad Sci U S A 115,E4255-E4263, doi:10.1073/pnas.1720941115 (2018).


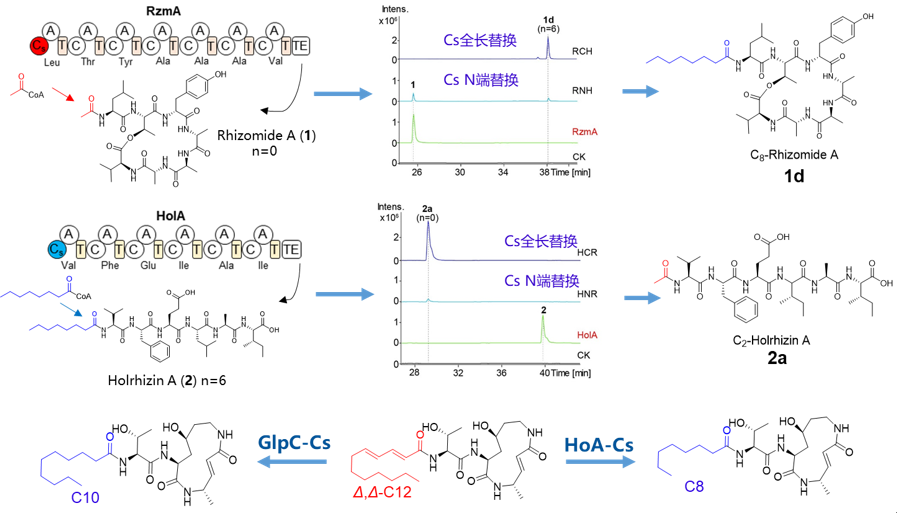
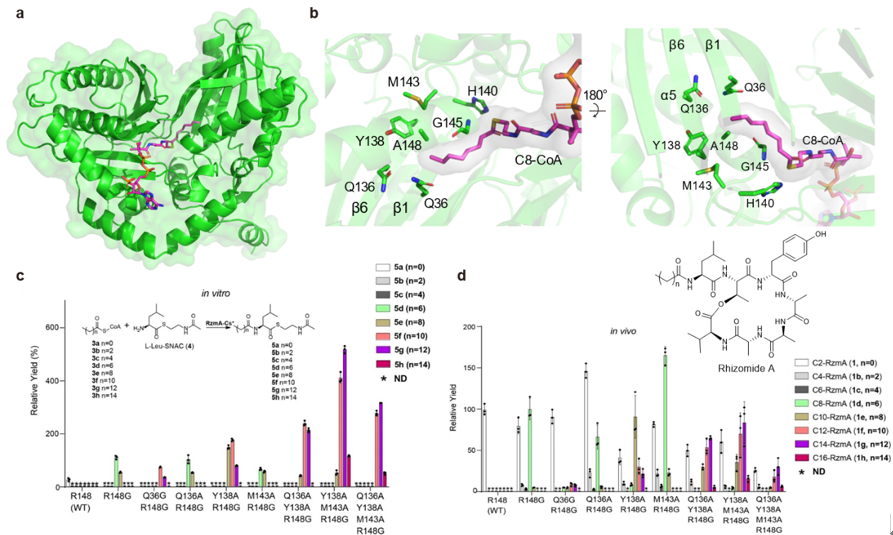
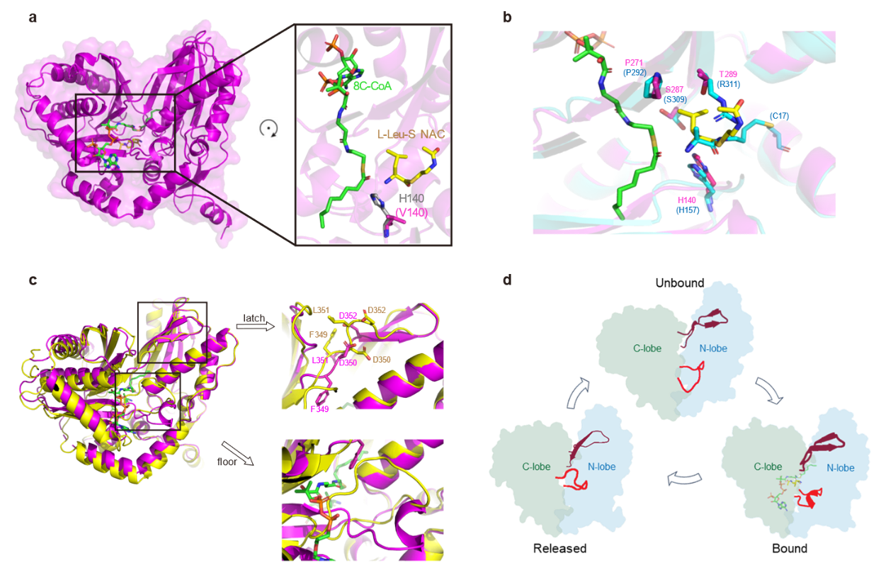

 药选址
药选址




