
以往认为中心法则中DNA主要发挥模板的作用而信使RNA(mRNA)则扮演着信息传递者的角色。mRNA表观修饰的发现拓展了mRNA的功能。在RNA水平上的转录后修饰可通过顺式或者反式机理调控基因表达,对细胞功能、细胞命运决定至关重要。尽管科学家们长时间以来了解RNA上存在转录后修饰,而这些修饰直到近年来才被认为从另一层面增加了基因表达调控的复杂性【1,2】。迄今为止,RNA上共发现有多于150种化学修饰【3】,包括m6A、m1A、pseudouridine、m5C等(图1)。这些修饰在“表观转录组”这一新颖且关键的层面调控基因的表达【4,5】。
m6A是mRNA内部含量最多,也是迄今研究最广泛的修饰【6】。m6A修饰最早发现于1974年,是哺乳动物mRNA内部最主要的修饰形式【1】,早期的研究工作表明m6A通常出现在(G/A)(m6A)C经典序列中【7,8】,其效应蛋白包括加上m6A修饰的“书写者”,擦掉m6A修饰的“擦除者”,以及识别m6A修饰的“识别者”(图1)。RNA 甲基化转移酶METTL3/14形成稳定的核心复合体作为“书写者”将mRNA上加上N6甲基化修饰【9,10】。去甲基化酶FTO【11】和ALKBH5【12】则作为“擦除者”将该修饰去除。“书写者”和“擦除者”动态调控了RNA m6A的修饰水平。m6A的识别蛋白含有YTH结构域,包括YTHDF1-3 , YTHDC1-2【13-17】,这些“识别者”识别RNA的m6A修饰并招募该RNA到特殊的蛋白复合体中,从而通过影响mRNA的代谢过程如剪接、出核、降解和翻译等调控基因表达,在发育,生理和病理过程中扮演重要角色【4,5,18】。
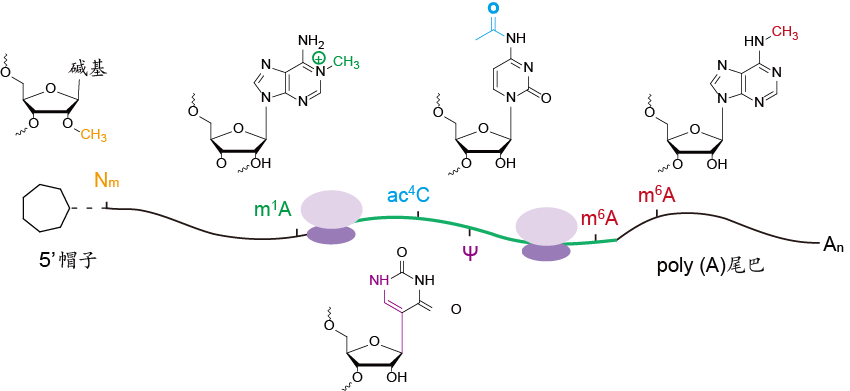
图1:mRNA常见的集中化学修饰,包括m6A、m1A、pseudo uridine(ψ)、m5C等。
最早的m6A修饰的全转录组图谱发表于2012年(m6A-Seq 或者 MeRIP-Seq),使用m6A抗体富集含有m6A的RNA片段,产生分辨率100~200 nt的图谱【19,20】。目前RNA m6A主流的测序方法依然是基于抗体的MeRIP,但该测序方法存在明显的缺点,包括分辨率低,无法定量,比较不同条件下m6A差异有局限性【21】,以及建库需要大量的RNA,不能应用于单细胞水平的研究等。近年来陆续开发的m6A测序方法(如Mazter-seq, m6A-REF-seq, DART-seq, m6A-SEAL, m6A-label-seq等)也只是部分解决了上述提及的局限性。
2022年3月14日,芝加哥大学何川教授联合复旦大学生物医学研究院/附属肿瘤医院胡璐璐研究员、美国希望之城陈建军教授和芝加哥大学陈梦洁教授在Nature Biotechnology杂志上发表了题为m6A RNA modifications are measured at single-base resolution across the mammalian transcriptome的文章,该研究开发的新方法m6A-SAC-Seq (Selective Allyl Chemical labeling and Sequencing)可直接标记m6A,能够覆盖几乎所有的m6A经典基序,并对捕获的m6A位点进行单碱基分辨率的定量分析(图2)。该方法不依赖于抗体,可以实现对微量RNA样本(30ng ribo- RNA)的m6A位点分析,可在单碱基分辨率水平追踪m6A分布和含量的动态变化。m6A-SAC-Seq技术是目前唯一可以广泛应用于各个生物学背景的方法,在基础生物学研究和临床应用中均有良好的前景。

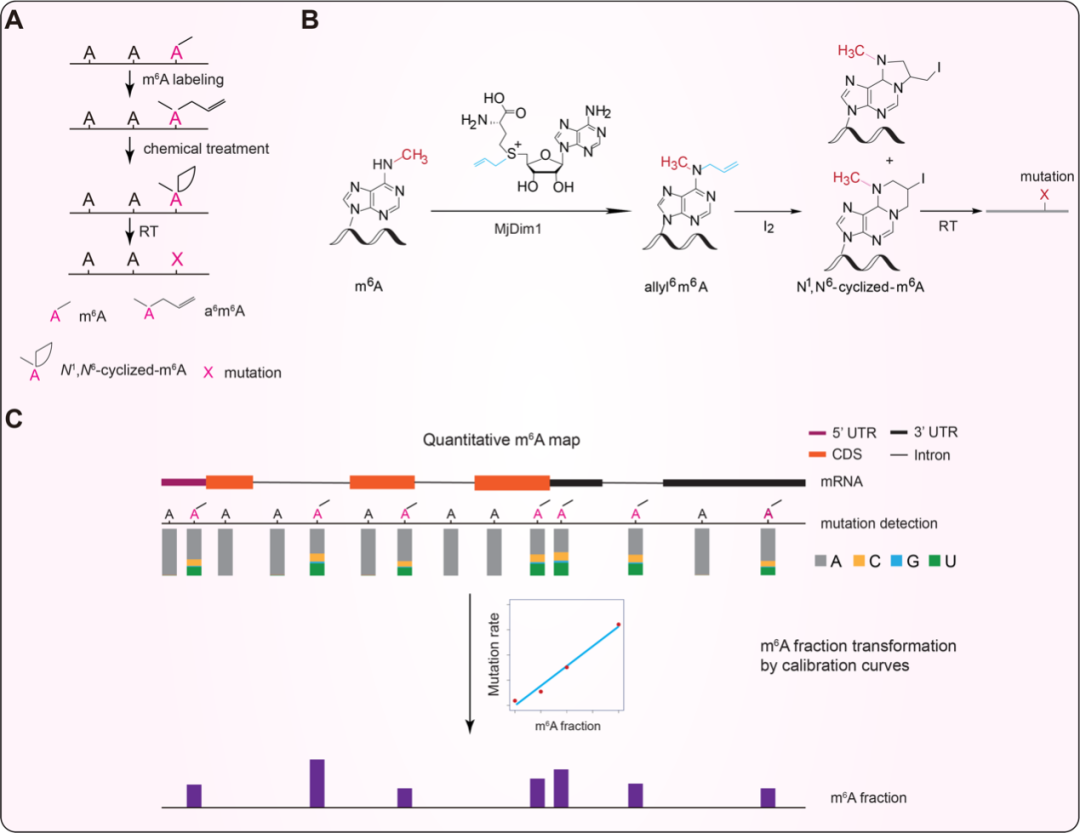
图2:m6A-SAC-Seq策略图。(A和B)使用特定的酶将m6A加上一个烯丙基的化学基团成为allyl6m6A,经过特殊的化学反应,发生成环反应,环化的allyl6m6A在逆转录时会被逆转录酶读成突变,根据突变的位点检测出m6A在转录组中的位置,通过突变率经过标准曲线换算获得m6A的准确的含量信息(C)。
研究人员使用m6A-SAC-seq技术在HeLa、HEK293和HepG2细胞系中各鉴定了大约一万个带有m6A含量信息的位点,并且这些位点在转录本上的分布集中在终止密码子附近、CDS和3’ UTR,符合之前的报道。通过结合分析RNA降解(decay)测序数据,研究人员还发现m6A含量和RNA半衰期呈现明显的负相关性,即m6A含量高的转录本更倾向于拥有短的更新周期(lifetime),这个结论进一步阐明了m6A修饰在调控mRNA更新过程(turnover)的规律。
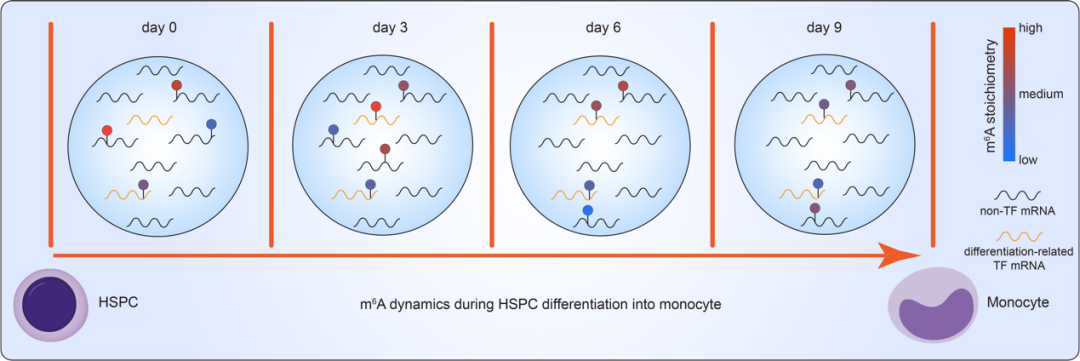
此外,研究人员还使用m6A-SAC-Seq技术成功绘制了人脐带血来源的造血干细胞(HSPC)分化为单核细胞(monocyte)的m6A含量动态图谱(图4)。在分化过程中,mRNA不同区域的m6A位点在分化过程中显示出其在mRNA 不同区域之间的大量重新分布和动态变化(图3A)。为进一步揭示m6A含量在该分化过程中的变化规律,研究人员根据mRNA在5' UTR、CDS、3' UTR和intron区域中的 m6A含量变化进行了两类分组,即动态(dynamic)修饰和稳定(stable)修饰。随后的功能富集分析表明,维持基本细胞存活功能,即与DNA修复、损伤、完整性和细胞周期的通路相关的基因往往会发生动态的m6A含量变化,而参与骨髓细胞活化、免疫反应、RNA剪接和分解代谢过程的基因的m6A含量更趋于稳定(图3B)。作者们经过分析确定了在连续两个时间点之间具有显著差异基因表达和m6A含量的候选转录本,并发现了这些转录本部分编码了对单核细胞发育调节至关重要的转录因子(TF)。例如,在第6天到第9天分化过程中,KLF4的CDS中m6A甲基化的含量随着转录本丰度提高而增加;在第0天到第3天分化过程中,ERG1、SPI1和CEBPA中3' UTR m6A甲基化的m6A含量减少,转录本丰度增加(图3C)。有趣地是,这些候选转录本还显著富集于主要转录因子(master TF,即对HSPC的发育过程至关重要的TF)的靶标集(图3D)。这些主要转录因子包括SP1、SPI1、NFKB1、RELA、STAT3和TP53等。

图3:A,人脐带血来源的造血干细胞分化为单核细胞生成过程中mRNA特征区域的整体m6A动态变化。B,造血干细胞分化过程中不同转录本区域中具有动态或稳定m6A修饰基因的功能富集富集分析热图。C,关键转录因子转录本m6A 含量变化和表达水平变化的示例图。D,m6A含量在CDS和3’ UTR变化显著以及基因表达变化显著的转录本在转录因子调控的靶标集中的富集热图。
该研究展示了m6A-SAC-Seq 技术在细胞分化(图4)、早期发育、神经元信号传导和临床样本中获得全转录组 m6A 含量变化的潜力。m6A-SAC-Seq 技术有望成为攻克当前定量 m6A 测序技术瓶颈并引领新生物学发现的“金标准”。
据悉,复旦大学胡璐璐生物医学研究院及附属肿瘤医院胡璐璐研究员、芝加哥大学何川课题组的刘顺博士、彭勇博士、葛睿琦博士研究生以及美国希望之城苏瑞研究员为本论文的共同第一作者,何川教授、陈建军教授、陈梦洁教授和胡璐璐研究员为共同通讯作者。
原文链接:
https://doi.org/10.1038/s41587-022-01243-z
2011年何川教授团队鉴定了第一个m6A去甲基化酶FTO【11】,拓展了RNA甲基化修饰研究新领域。m6A修饰参与调控RNA代谢加工(如剪接、出核、降解和翻译等)及基因表达和发育、生理和病理过程。
m6A测序技术的发展对推进RNA甲基化规律前沿和应用研究至关重要,其中应用最广泛的是基于m6A抗体免疫共沉淀的MeRIP-seq技术,近年来多种基于不同原理的m6A测序技术(如Mazter-seq, m6A-REF-seq, DART-seq, m6A-SEAL, m6A-label-seq等)被研发,极大的拓展了m6A功能研究的维度,这些新技术解决了部分MeRIP-seq技术在样本量、分辨率、定量化等方面的局限性【19】。
m6A-SAC-Seq技术攻克了m6A测序关键性技术瓶颈,为m6A领域的科研活动提供了单碱基定量的实用工具。m6A-SAC-Seq技术原理是将m6A通过特定的酶加上一个烯丙基基团,通过化学处理产生环化的具有空间位阻的基团,从而在逆转录的时候通过逆转录酶引入突变。m6A-SAC-Seq能够捕获大部分经典的位于Gm6AC motif的m6A位点。。应用m6A-SAC-Seq技术,绘制了人脐带血来源的造血干细胞(HSPC)分化为单核细胞(monocyte)的m6A动态图谱。
m6A-SAC-Seq技术使用酶标记法直接标记m6A修饰,而不是条件更苛刻的化学处理方法,也不需要通过反向扣除,极大的降低了背景噪音,提升了m6A位点的准确性,m6A-SAC-Seq技术的方法,具有样本低起始量的技术优势,在基础研究领域和临床应用中检测全转录组 m6A 含量变化方面都具有广泛的应用前景。
最后,复旦大学生物医学研究院和附属肿瘤医院的胡璐璐研究员课题组当前的研究方向是单细胞表观基因组和表观转录组,空间表观基因组和表观转录组新技术的开发,实验室现在招收博士后,生物化学与分子生物学,细胞生物学和生物信息学方向优先。

参考文献
1. Desrosiers, R., Friderici, K. & Rottman, F. Identification of methylated nucleosides in messenger RNA from Novikoff hepatoma cells. Proc Natl Acad Sci U S A 71, 3971-3975 (1974).
2. Rottman, F., Shatkin, A.J. & Perry, R.P. Sequences containing methylated nucleotides at the 5' termini of messenger RNAs: possible implications for processing. Cell 3, 197-199 (1974).
3. Helm, M. & Motorin, Y. Detecting RNA modifications in the epitranscriptome: predict and validate. Nat Rev Genet 18, 275-291 (2017).
4. Frye, M., Harada, B.T., Behm, M. & He, C. RNA modifications modulate gene expression during development. Science 361, 1346-1349 (2018).
5. Roundtree, I.A., Evans, M.E., Pan, T. & He, C. Dynamic RNA Modifications in Gene Expression Regulation. Cell 169, 1187-1200 (2017).
6. Shi, H., Wei, J. & He, C. Where, When, and How: Context-Dependent Functions of RNA Methylation Writers, Readers, and Erasers. Mol Cell 74, 640-650 (2019).
7. Schibler, U., Kelley, D.E. & Perry, R.P. Comparison of methylated sequences in messenger RNA and heterogeneous nuclear RNA from mouse L cells. J Mol Biol 115, 695-714 (1977).
8. Wei, C.M. & Moss, B. Nucleotide sequences at the N6-methyladenosine sites of HeLa cell messenger ribonucleic acid. Biochemistry 16, 1672-1676 (1977).
9. Bokar, J.A., Shambaugh, M.E., Polayes, D., Matera, A.G. & Rottman, F.M. Purification and cDNA cloning of the AdoMet-binding subunit of the human mRNA (N6-adenosine)-methyltransferase. RNA 3, 1233-1247 (1997).
10. Liu, J. et al. A METTL3-METTL14 complex mediates mammalian nuclear RNA N6-adenosine methylation. Nat Chem Biol 10, 93-95 (2014).
11. Jia, G. et al. N6-methyladenosine in nuclear RNA is a major substrate of the obesity-associated FTO. Nat Chem Biol 7, 885-887 (2011).
12. Zheng, G. et al. ALKBH5 is a mammalian RNA demethylase that impacts RNA metabolism and mouse fertility. Mol Cell 49, 18-29 (2013).
13. Wang, X. et al. N6-methyladenosine-dependent regulation of messenger RNA stability. Nature 505, 117-120 (2014).
14. Wang, X. et al. N(6)-methyladenosine Modulates Messenger RNA Translation Efficiency. Cell 161, 1388-1399 (2015).
15. Shi, H. et al. YTHDF3 facilitates translation and decay of N(6)-methyladenosine-modified RNA. Cell Res 27, 315-328 (2017).
16. Roundtree, I.A. et al. YTHDC1 mediates nuclear export of N(6)-methyladenosine methylated mRNAs. eLife 6 (2017).
17. Hsu, P.J. et al. Ythdc2 is an N(6)-methyladenosine binding protein that regulates mammalian spermatogenesis. Cell Res 27, 1115-1127 (2017).
18. Yang, Y., Hsu, P.J., Chen, Y.S. & Yang, Y.G. Dynamic transcriptomic m(6)A decoration: writers, erasers, readers and functions in RNA metabolism. Cell Res 28, 616-624 (2018).
19. Huan Yao, Ying Yang, Yun-Gui Yang. scDART-Seq: Mapping m6A at single cell level, Mol Cell, 2022, 82(4):713-715. (Preview)
(可上下滑动阅览)
转载须知
【非原创文章】本文著作权归文章作者所有,欢迎个人转发分享,未经允许禁止转载,作者拥有所有法定权利,违者必究。




 个人中心
个人中心
 我是园区
我是园区




