Nature | 可爱龙实验室揭示CRISPR-Cas系统Prespacer方向性整合机制
收藏
关键词:
Nature揭示PreNat
资讯来源:BioArt + 订阅账号
发布时间:
2021-09-30

责编 | 兮
CRISPR-Cas系统是目前应用最广泛的基因编辑工具,其起源是来自于细菌和古细菌中针对噬菌体,病毒或其他外源入侵核酸的一种获得性免疫系统。其最大亮点是其可编程的指引RNA
(guide RNA)
介导效应器Cas蛋白
(如Cas9, Cas12, Cas12, Cascade等等)
可以对任意指定的核酸序列
(DNA或RNA)
进行切割或编辑
【1】
。从首次对其功能报道至今不过10年左右,然而因其巨大的应用价值,获得了令人惊讶的速度蓬勃发展,其发现者Jennifer Doudna, Emmanuelle Charpentier已经于2020年分享诺贝尔化学奖。
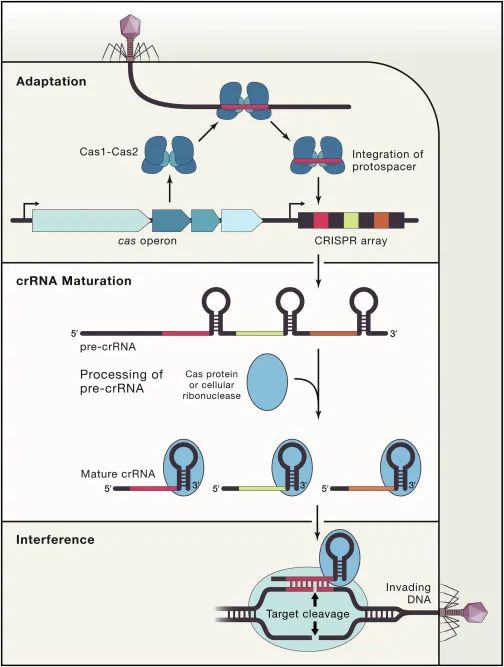
CRISPR-Cas系统的工作分为三步,如下图所示
【1】
。首先是CRISPR系统整合酶Cas1-Cas2复合物捕捉外源入侵DNA序列
(如图中的噬菌体)
,再将其整合到CRISPR Array当中去,该步骤被称为Adaptation。然后CRISPR Array会被转录为RNA序列
(pre-RNA)
,经过相关的Cas蛋白加工成为较短的、成熟的指引RNA
(guide RNA)
序列,再与效应器
(Cas9, Cas12, Cas12, Cascade等)
结合成为具有靶向功能的效应器复合物。最后当存在与guide RNA相同序列的外源性DNA再次入侵时,效应器会立刻启动靶向,从而对入侵物进行切割清除。
其中第一步的Cas1-Cas2捕捉和整合外源DNA的Adaptation过程已经有比较多的研究报道
【2-6】
。这其中包括中国科学院生物物理所王艳丽研究员实验室2015年发表于Cell的工作报道了Cas1-Cas2如何识别PAM序列
【2】
。稍后时间Jenifer实验室通过Nature长文也阐述了类似观点。之后于2017年几乎同期发表的Nature和Science分别报道了可爱龙研究组和Jennifer Doudna研究组的Cas1-Cas2-Prespacer-Target DNA复合物结构,至此基本上讲清Cas1-Cas2复合物是如何捕捉外源性的DNA
(Prespacer)
并如何整合进入CRISPR Array
(Target DNA)
的分子机制。然而有一个比较关键的问题:Cas1-Cas2整合Prespacer进入CRISPR Array是具有方向性的,比如在Type I系统之中,PAM链一定是滞后整合进入Repeat-Spacer端
【7】
(如下图所示)
,然而其具体机制目前不是很明确。
2021年9月29日, Nature以长文形式报道了来自美国康奈尔大学可爱龙实验室题为
Mechanism for Cas4-assisted directional spacer acquisition in CRISPR-Cas
的研究成果。该论文
首次阐述了CRISPR-Cas系统中Cas4如何识别PAM并协助Cas1-Cas2捕捉Prespacer,以及如何调控Prespacer进行方向性整合的分子机制。
2018年以来陆续有众多研究工作开始报道,Cas4对Prespacer的选择、加工以及方向性整合具有至关重要的作用
【8-10】
。不过这些工作基本上都是从体内in vivo的进行的功能阐述,无法解释Cas4这些行为的具体的机制。此外需要注意的是,Cas4首次的单亚基晶体结构报道实际上可追溯到2012年左右,然而这时期的对Cas4功能阐述观点不一,存在很多争议,因此有关Cas4的问题依然是一团疑云。Cas4研究的障碍在于Cas4是一种Fe-S铁硫簇家族核酸酶,其Fe-S对氧气异常敏感从而导致蛋白迅速变性沉淀,此外其底物也不明确,因而
体外对其复合物组装工作比较难开展。2019年荷兰Delft University of Technology的Stan J J Brouns团队发现一种高效的来源于Geobacter sulfurreducens的GsCas4-Cas1融
合系统
【10】
,该融合系统的发现可能对Cas4的稳定性提高有一定帮助。
基于此背景,可爱龙教授带领其实验室胡纯一博士等人,联合Stan J J Brouns
团队,使用GsCas4-Cas1融合系统,在优化了很多纯化条件之后,最终在厌氧条件下获得了性质稳定、均一的Cas4-Cas1蛋白。之后尝试组装Cas4-Cas1-Cas2-Prespacer_Dual PAM复合物,其团队通过筛选底物,获得了高度均一的复合物,并且使用冷冻电镜解析了其3.0埃分辨率的复合物结构。结构首次揭示了在此系统之中是由Cas4识别PAM序列,而非以往报道的由Cas1负责识别PAM
(如下图所示)
。同时,他们发现新鲜纯化出来的样品展现出的是异常精确的特异性切割——Cas4只会以内切酶形式切割PAM序列,然而当样品经过氧化后,Cas4的Fe-S簇被破坏,Cas4的活性就会杂乱无章,对序列没有特异性,同时会展现出内切酶和外切酶活性。这个结论,解释了以往关于Cas4酶活不同性质的报道,一定程度上解决了这些争议问题。
之后团队继续解析了Cas4-Cas1-Cas2-Prespacer_PAM/Non PAM复合物结构。发现如果Prespacer的3' 端不存在PAM序列的话,Cas4会释放该末端,并呈递给宿主细胞的核酸酶。后续的生化试验也证明了核酸酶ExoI能迅速切割Non-PAM末端,而PAM端被Cas4紧紧保护从而无法被切割。这揭示了PAM端滞后整合的分子基础。
(如下图所示)
不过这些结论只能解释Cas4识别PAM协助Cas1-Cas2捕捉外源DNA的分子机制,无法完全解释方向性整合问题。后来可爱龙团队通过高分辨率结构得知完整的复合物中Cas4虽然结合底物以及金属离子,然后其并未切割PAM而只是识别结合。因此猜测必定还存在某种机制激活Cas4酶活,再对PAM进行切割加工。在此基础之上,团队继续解析了一系列Cas4-Cas1-Cas2-Prespacer-Target DNA半整合以及全整合结构,通过结构和生化分析,揭示了在Non-PAM端被ExoI快速加工后会迅速被整合进入CRISPR Array的Leader-Repeat端,从而形成半整合状态。在此半整合状态下,一段突出的DNA序列会结合Cas4-Cas1蛋白一块正电荷富集区,然后推动Cas4高效切割PAM,切割完成后Cas4会将末端传递给Cas1整合酶活性中心完成全整合过程
(如下图所示)
。
该项研究首次揭示了CRISPR-Cas系统中Cas4通过识别PAM序列协助整合酶Cas1-Cas2捕捉Prespacer的分子机制,此外通过一些结构和生化手段揭示了Cas4介导的Prespacer方向性整合的详细分子机制。
最后值得称道的是,可爱龙教授持续深耕于CRISPR-Cas领域,发表于今天的Nature工作是其继2016年Nature报道Type I-E系统Cascade识别PAM机制;2017年7月Cell发表I-E系统Cascade/R-loop分子机制以及同年9月Nature报道II-A系统Cas1-Cas2整合Prespacer进入CRISPR Array的分子机制;2018年Science报道Cascade/R-loop招募Cas3的分子基础;2020年NSMB报道使用单分子手段实时观察Cas1-Cas2整合行为之后,在该领域又一项具有里程碑式意义的研究。据悉,可爱龙实验室有结构生物学和细胞生物学博士后职位提供。
https://jinshuju.net/f/ZqXwZt
或扫描二维码
投递简历
https://www.nature.com/articles/s41586-021-03951-z
1 Hille, F. et al. The Biology of CRISPR-Cas: Backward and Forward. Cell 172, 1239-1259, doi:10.1016/j.cell.2017.11.032 (2018).
2 Wang, J. et al. Structural and Mechanistic Basis of PAM-Dependent Spacer Acquisition in CRISPR-Cas Systems. Cell 163, 840-853, doi:10.1016/j.cell.2015.10.008 (2015).
3 Nunez, J. K. et al. Cas1-Cas2 complex formation mediates spacer acquisition during CRISPR-Cas adaptive immunity. Nature structural & molecular biology 21, 528-534, doi:10.1038/nsmb.2820 (2014).
4 Xiao, Y., Ng, S., Nam, K. H. & Ke, A. How type II CRISPR-Cas establish immunity through Cas1-Cas2-mediated spacer integration. Nature 550, 137-141, doi:10.1038/nature24020 (2017).
5 Wright, A. V. et al. Structures of the CRISPR genome integration complex. Science 357, 1113-1118, doi:10.1126/science.aao0679 (2017).
6 Nunez, J. K., Harrington, L. B., Kranzusch, P. J., Engelman, A. N. & Doudna, J. A. Foreign DNA capture during CRISPR-Cas adaptive immunity. Nature 527, 535-538, doi:10.1038/nature15760 (2015).
7 Jackson, S. A. et al. CRISPR-Cas: Adapting to change. Science 356, doi:10.1126/science.aal5056 (2017).
8 Lee, H., Zhou, Y., Taylor, D. W. & Sashital, D. G. Cas4-Dependent Prespacer Processing Ensures High-Fidelity Programming of CRISPR Arrays. Molecular cell 70, 48-59 e45, doi:10.1016/j.molcel.2018.03.003 (2018).
9 Shiimori, M., Garrett, S. C., Graveley, B. R. & Terns, M. P. Cas4 Nucleases Define the PAM, Length, and Orientation of DNA Fragments Integrated at CRISPR Loci. Molecular cell 70, 814-824 e816, doi:10.1016/j.molcel.2018.05.002 (2018).
10 Almendros, C., Nobrega, F. L., McKenzie, R. E. & Brouns, S. J. J. Cas4-Cas1 fusions drive efficient PAM selection and control CRISPR adaptation. Nucleic acids research 47, 5223-5230, doi:10.1093/nar/gkz217 (2019).
【非原创文章】本文著作权归文章作者所有,欢迎个人转发分享,未经允许禁止转载,作者拥有所有法定权利,违者必究。










 药选址
药选址









