
责编 | 兮
我们的生命由单个受精卵细胞形成时开始。在母体子宫内,受精卵通过亿万次的细胞分裂而发育成多细胞个体。在每次的细胞分裂中,编码遗传信息的基因组DNA都会被准确复制。每个细胞携带的DNA总长度约为两米,其通过折叠、整合成为23对染色体。在人的一生中
(约70年)
,人体将合成接近一光年长度的DNA
(1016米)
。在复制的起始过程中,首先需要利用解旋酶将双链DNA分解成两条单链DNA,其后DNA聚合酶以此为模板,合成两条新的互补配对的双螺旋DNA。该复制过程受到严格的调控,任何调控的紊乱都可能导致遗传疾病或形成肿瘤等严重后果。
自1953年James WATSON和Francis CRICK解析DNA双螺旋结构以来,DNA双链最初是如何被解旋并打开,一直是个未解之谜
。1983年,本研究的合作者,康奈尔大学的戴碧瓘教授在酿酒酵母中发现了真核生物的DNA解旋酶基因——MCM
【1】
。6个MCM基因编码的MCM2-7 蛋白
(Minichromosome Maintenance 2-7,微小染色体维持蛋白2-7)
形成一个六亚基的环状复合体。在细胞周期的G1期
(又称准备期)
, 在ORC1-6
(Origin Recognition Complex 1-6,复制起始点识别复合体1-6)
、CDC6和CDT1的帮助下, MCM2-7复合物被组装到染色体上的成千上万个复制原点,形成环绕双链DNA的MCM2-7 Double Hexamer
(MCM2-7 DH, MCM2-7双六聚体)
。进入复制期
(S期)
后,一部分MCM2-7 DH被激活并连同其他复制因子一起,最终形成两个复制体
(Replisome)
开始进行双向DNA复制。因此,MCM2-7 DH又被称为pre-Replication Complex
(pre-RC,复制前始复合物)
。一直以来,MCM2-7 DH被认为具有直接解旋和打开双链DNA的能力,但其作用机制尚不清楚。
为解开这个疑团,香港大学翟元梁教授领导的团队,使用冷冻电镜技术
(Cryo-EM)
研究与复制起始过程相关联的大分子复合物的高分辨蛋白质结构,从而了解与MCM2-7蛋白复合体相关的DNA解链的分子机制,并取得了多项突破性成果
【2-5】
。在2015年,翟元梁/戴碧瓘团队与北京
大学生命科学学院的高宁教授一同首次解析了从酿酒酵母里提纯的内源MCM2-7 DH冷冻电镜结构,相关成果发表于Nature杂志
(Structure of the eukaryotic MCM complex at 3.8 Å, DOI: 10.1038/nature14685)
。然而,在该结构中,复制原点的DNA非常不稳定,无法提供MCM2-7 DH与双螺旋DNA相结合状态的信息。
2023年1月5日,香港大学翟元梁教授联同香港科技大学党尚宇教授、香港科技大学/康奈尔大学戴碧瓘教授和法国居里研究所陈春龙教授,在 Cell杂志上发表标题为
The Human Pre-replication Complex is an Open Complex
的研究论文。相关研究人员
成功从HeLa细胞中纯化得到人内源MCM2-7 DH-DNA复合物,并解析出2.59埃高分辨率冷冻电镜结构,揭示了人细胞DNA复制起始新机制。
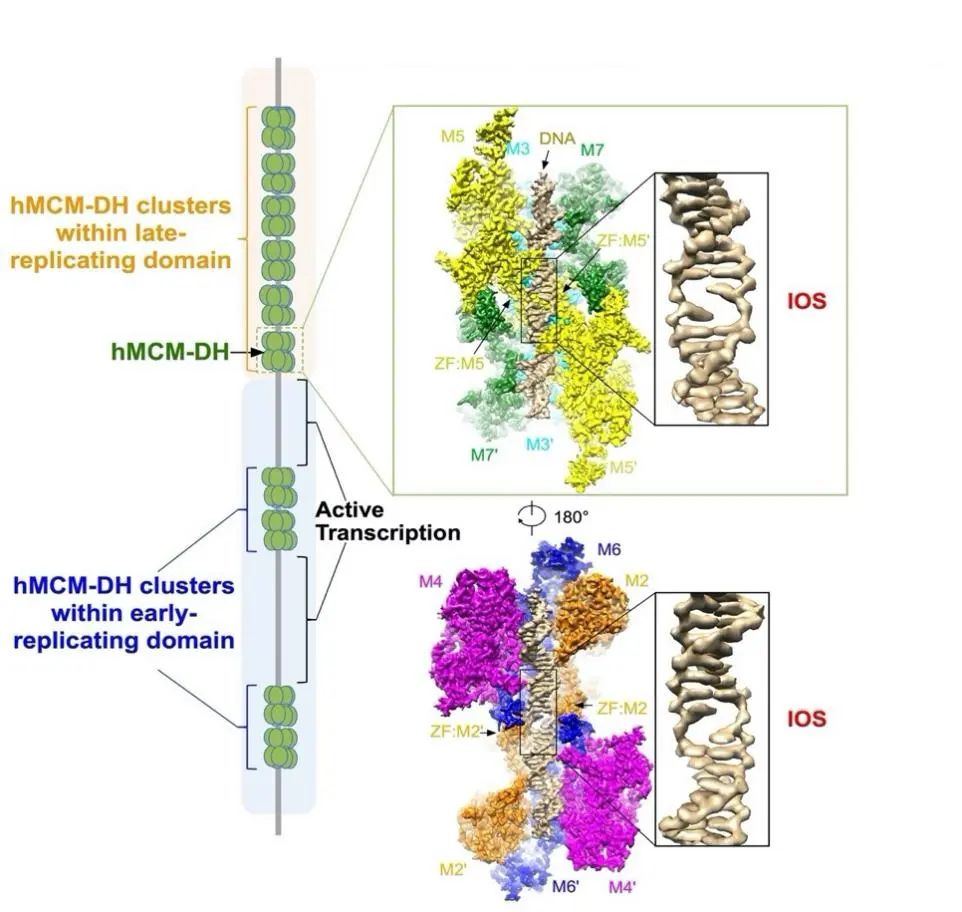
该结构清晰地展示了MCM2-7 DH如何与DNA双链结合,并将位于两个六聚体结合处的双链DNA解旋,形成一个初始打开结构(Initial Open Structure, IOS)
(图1)
,从而使得MCM2-7 DH牢固结合在初始打开的复制起始位点DNA上。为形成该IOS,MCM-DH在MCM2-7六聚体结合处,通过MCM锌指结构域
(ZF)
构成的两个互相交错的锌指环以及临近的结构单元
(motif)
,压缩MCM中心通道孔径到仅有13埃;同时两个MCM2-7通过反向协调旋转,拉伸和解旋双链DNA。结果,复制起始位点半个双螺旋长度的双链DNA稳定性被降低,而位于正中心的一对碱基被完全打开。这些打开的IOS刚好被附近来自MCM2和MCM5的两对锌指结构域结合,使得MCM-DH复合体蛋白能够牢固抓住DNA。研究人员进一步发现,如果IOS被破坏或干扰,所有的MCM2-7 DH将无法稳定结合在DNA上,从而完全抑制DNA复制的起始。
通过提取人HeLa细胞系的MCM2-7 DH结合的DNA并进行高通量测序,研究人员获得了首个人类MCM-DH在整个基因组结合位点接近单个核苷酸分辨率的图谱。生物信息分析结果显示,在早期复制区域,MCM2-7 DH的结合位点形成大小不同分布的簇群
(clusters)
,这些簇群与之前在HeLa细胞系通过冈崎片段测序
(OK-seq)
【6】
在群体水平测得的复制起始区域,以及通过Optical Replication Mapping
(ORM)
单分子成像技术
(详见BioArt报道:
鉴定的复制起始富集区
【7】
高度吻合。重要的是,早期的MCM2-7 DH簇群富集于基因间隔区与活跃的信使RNA
(mRNA)
转录区域互斥,该结果支持了转录塑造DNA复制图谱的观点
【8】
。但是,
在晚期复制区域内,MCM2-7 DH有着更高的密度和更广泛的分布,这一发现也说明DNA复制起始效率并非由特定位点的MCM2-7 DH数量来决定
【9,10】(图1)
。
图1. 人源MCM双六聚体(hMCM-DH)结合复制原点产生的DNA初始解开结构(IOS)
长期以来,DNA复制程序是多种癌症治疗药物的作用靶点。但由于正常细胞和癌症细胞在增殖时都需要进行DNA复制,导致这些药物会无差别地杀死所有进入复制过程的细胞,因此往往有较大的毒性。如何提高化疗药物作用的针对性,一直是研发抗癌药物的重要考虑因素。在现阶段,一个理想方案是通过抑制DNA复制起始,使正常细胞滞留在G1期或者在G0期静止;在此情况下,癌症细胞则因其调控机制的紊乱,无法像正常细胞一样被保护在相应的细胞周期,从而被诱导进入细胞凋亡。因此,阻断细胞复制起始,有望成为一种全新的、有效的、高度特异针对癌症细胞的抗癌疗法。
该项研究揭示的人源复制前始复合物(pre-RC)的蛋白结构和工作机制,为以MCM2-7复合体为靶点的无副作用抗癌药物开发提供了设计思路和重要的结构基础。
香港大学翟元梁教授、香港科技大学党尚宇教授、香港科技大学/康奈尔大学戴碧瓘教授、法国居里研究所陈春龙教授为论文共同通讯作者。香港大学博士研究生李健、香港科技大学董江清博士、法国居里研究所王纬韬博士、香港科技大学余大启博士为论文共同第一作者。
香港大学的翟元梁课题组,长期招聘优秀博士后和博士生,有意者请参看BIOART招聘:
https://zhai95.wixsite.com/mysite-1
法国居里研
究所陈春龙课题组有多个博士后职位
(wet和dry labs)
正在招聘中,详细信息见:
https://institut-curie.org/offre-emploi/postdoctoral-position-genomics-mf-0 。
香港科技大学党尚宇课题组,长期招聘优秀博士后和博士生,课题组详细信息请参阅:
https://facultyprofiles.hkust.edu.hk/profiles.php?profile=shangyu-dang-sdang。
原文链接:
https://doi.org/10.1016/j.cell.2022.12.008
制版人:十一
1 Maine, G. T., Sinha, P. & Tye, B. K. Mutants of S. cerevisiae defective in the maintenance of minichromosomes. Genetics 106, 365-385, doi:10.1093/genetics/106.3.365 (1984).
2 Li, N. et al. Structure of the eukaryotic MCM complex at 3.8 A. Nature 524, 186-191, doi:10.1038/nature14685 (2015).
3 Zhai, Y. et al. Open-ringed structure of the Cdt1-Mcm2-7 complex as a precursor of the MCM double hexamer. Nat Struct Mol Biol 24, 300-308, doi:10.1038/nsmb.3374 (2017).
4 Cheng, J. et al. Structural Insight into the MCM double hexamer activation by Dbf4-Cdc7 kinase. Nat Commun 13, 1396, doi:10.1038/s41467-022-29070-5 (2022).
5 Li, N. et al. Structure of the origin recognition complex bound to DNA replication origin. Nature 559, 217-222, doi:10.1038/s41586-018-0293-x (2018).
6 Petryk, N. et al. Replication landscape of the human genome. Nat Commun 7, 10208, doi:10.1038/ncomms10208 (2016).
7 Wang, W. et al. Genome-wide mapping of human DNA replication by optical replication mapping supports a stochastic model of eukaryotic replication. Mol Cell 81, 2975-2988 e2976, doi:10.1016/j.molcel.2021.05.024 (2021).
8 Kirstein, N. et al. Human ORC/MCM density is low in active genes and correlates with replication time but does not delimit initiation zones. eLife 10, doi:10.7554/eLife.62161 (2021).
9 Hyrien, O. How MCM loading and spreading specify eukaryotic DNA replication initiation sites. F1000Res 5, doi:10.12688/f1000research.9008.1 (2016).
10 Das, S. P. et al. Replication timing is regulated by the number of MCMs loaded at origins. Genome Res 25, 1886-1892, doi:10.1101/gr.195305.115 (2015).
【非原创文章】本文著作权归文章作者所有,欢迎个人转发分享,未经作者的允许禁止转载,作者拥有所有法定权利,违者必究。




 药选址
药选址


