
二代测序技术已经走进“千家万户”,它可以帮助老师们对于自己的样本进行基因组层面的分析。如何有效的构建自己的文库,也成为每个实验室需要考虑的问题,小编在这儿为您总结了一些有关文库构建的知识,来帮助您更好的理解DNA建库的实验。
1.关于Input DNA
DNA质量
Input DNA的质量、片段大小以及浓度对于文库是否可以成功构建是至关重要的,这就需要对于不同类型的样本&不同的应用开发不同的protocol来构建有效的文库。同时,对于明确的样本类型以及应用,也要尽可能保证input DNA浓度上、片段大小以及质量的一致性。
根据不同样本类型以及不同的应用,可以选择多种方式对于input DNA做均一化。可以选择的分析方式从便宜且简单的方式到昂贵复杂的流程,通常包括:使用spectrophotometric 或者fluorometric (如PicoGreen®)来分析浓度;确定片段大小或者片段选择的方法;通过qPCR的方式来确定DNA的质量或者浓度(如KAPA Human Genomic DNA Quantification and QC Kit)。
DNA溶解Buffer组成
末端修复的酶反应需要游离的Mg2+,所以input DNA应该溶解于水中或者低盐的缓冲液中,几乎不包含或者不包含如EDTA的金属离子螯合剂。
片段化
Input DNA的大小应与选择的应用方向有关,您可以选择不同的片段大小来对目标基因组进行组装或者分析。通常DNA片段化的方式有ultrasonic, mechanical, 或者enzymatic的方法。最常见的方式是使用covaris来进行片段化,打断的片段大小较为集中重复性好;近年来开始使用酶切的方式进行DNA的片段化,通过控制酶切温度以及时间来实现DNA打断的不同大小,并且可以使用自动化的系统。KAPA Hyperplus系统使用了酶切打断的方案,实现了超高的DNA转换效率。
2. Master mix稳定性
Master mix的稳定性对于自动化实验非常重要。我们可以对于试剂的稳定性进行测试,来帮助我们合理的设置自动化程序。如下图为对于KAPA HTP试剂盒进行的master mix稳定性测试(从end repair 到adapter Ligation),setup 1(灰色)和setup 2(白色)分别为完整的master mix和没有加水的master mix;分别测试了4度保存2、4、7天,或者经历1、2、3次冻融的过程。
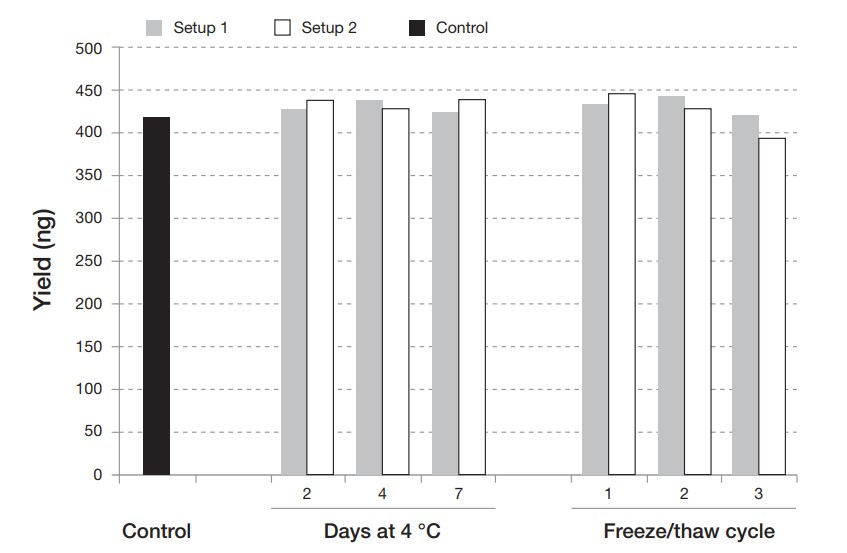
图:KAPA HTP试剂盒进行的master mix稳定性测试
如果将KAPA Hyper使用自动化工作站,酶以及buffer应混合成master mix而不应单独吸取;Master mix可以室温保存24小时以内,不需要冷却。由于KAPA HiFi HotStart DNA Polymerase极强的3-〉5外切酶的活性,PCR master mix与引物不能长时间放在一起,扩增体系需要新鲜配制。
3. Adapter以及连接后纯化
通常,adapter的连接效率可以耐受一定范围的adapter与insert的分子比,所以在一定的起始量上不需要对于每个单独样本调整adapter的浓度。我们可以根据下图的公式来计算input DNA的分子数。
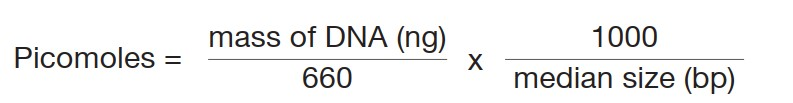
不同浓度adapter对于文库产量的影响
过高的adapter浓度可以导致连接后的磁珠纯化之后还有adapter以及adapter-dimer的残留。相反的,过低的adapter浓度降低了连接效率导致过多的分子末端只有单端连接上接头。我们可以通过QPCR的方法如KAPA Library Quantification Kit对于连接后的有效文库进行准确的定量。
关于Y型接头
下图所示为典型的Y型接头的2100电泳图(1μl,0.6μM), Y型接头单链长度约60bp,在电泳图中有两个峰,推测是由于两条adapter单链臂相互作用。
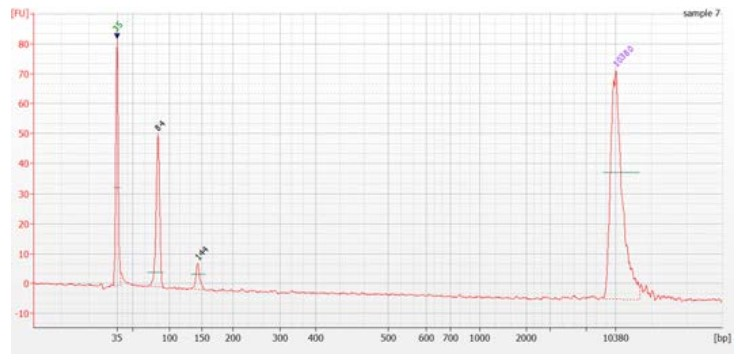
图:Y型接头的2100电泳图
Adapter dimer的去除
连接反应完成后通常使用0.8x磁珠(KAPA Beads or AMPure XP)去除残留的adapter,要确保磁珠平衡至室温并且在使用之前完全重悬;要一直使用新鲜配制的80%乙醇,并且在清洗过程中磁珠要完全浸没在乙醇中。
4.文库扩增
多数的文库构建工作流程都会包含1次或者多次的PCR扩增步骤,来增加连接好接头的DNA片段的量,或者增加功能性序列如index等。尽管KAPA HiFi HotStart DNA Polymerase有着非常低的扩增偏好以及高的保真度,但是过度扩增依然需要避免,过度扩增会带来duplicate reads增加&覆盖深度的不均一&嵌合文库&核酸替换等。我们应该在方法学开发的时候根据项目内容,对不同的input量和PCR循环数来评估文库的扩增。
下表为不同起始量的模板(使用QPCR方法KAPA Library Quantification Kit定量)以及PCR循环数所得到的扩增产量(浓度使用KAPA Library Quantification Kit计算,平均长度550bp)。
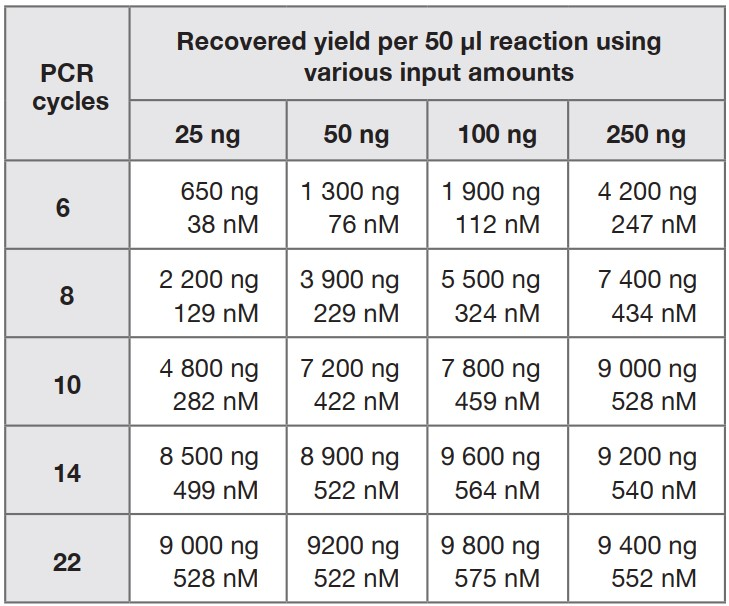
图:不同起始量的模板以及PCR循环数所得到的扩增产量
使用QPCR或者NanoDrop的方法评估文库产量有很好的一致性,50μl的体系所达到的平台期在8-10μg的扩增产物。相反,如果使用PicoGreen® 或者Bioanalyzer的方法来分析则会出现随着PCR循环数增加产量下降的现象,这是由于随着PCR循环的进行,引物以及dNTPs全部消耗,单个分子不再能通过引物退火延伸转换为双链DNA,而形成了所谓的“daisy-chains”,而影响到了PicoGreen® 或者Bioanalyzer的定量检测。
下图所示为25ng DNA分别扩增6 cycles (a), 8 cycles (b), 12 cycles (c),14 cycles (d) or 22 cycles (e),1x磁珠纯化所得到的结果。
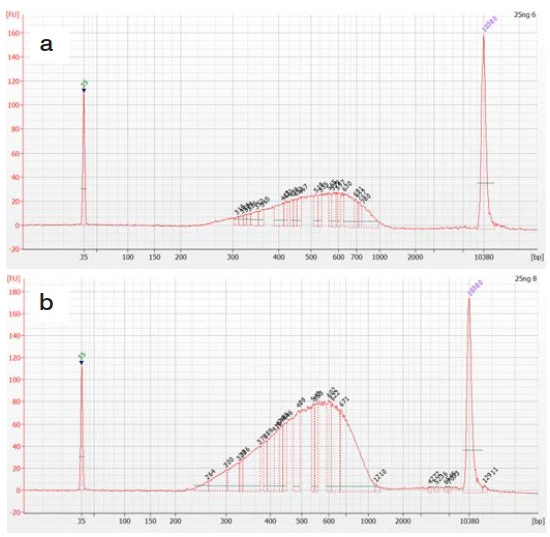
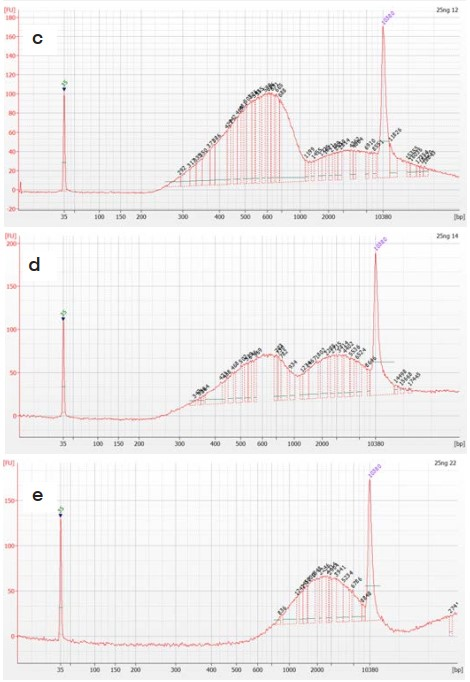
二代测序的文库制备已经越来越容易操作,但是仍然需要老师们对于自己项目的样本以及建库实验中每个步骤有着深刻的理解,从而获得有效的数据进行下游分析。如果有任何建库相关问题,非常欢迎与我们一起交流讨论。
参考资料:
“High-Throughput NGS Library Preparation Technical Guide” – Roche Diagnostics
*仅供生命科学研究,不用于临床诊断
MC-CN-01554 有效期2024年3月31日
目前已有500+行业精英加入基因俱乐部
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()




 个人中心
个人中心
 我是园区
我是园区





