
近年来,长读长测序技术等新技术的发展,使得完全解析和组装人类基因组的两种单倍型成为可能。与常用的长读长基因组组装相比,分阶段人类基因组组装方法更容易发现变异,其中结构变异(SVs)— 长度超过50bp的倒置、缺失、重复和插入的检测灵敏度提高最大,通常可检测到超过20,000个SVs。
近期,人类基因组结构变异国际合作联合会(HGSVC)开发了一种阶段性基因组组装方法,该方法整合了PacBio长读长全基因组测序(WGS)技术和链特异性测序(Strand-Seq)技术,可产生完全分阶段的二倍体基因组组装,而无需依赖亲子三人组数据(parent–child trio data)。这种阶段性组装可帮助我们获得更完整的人类基因组变异序列解析。
基于此,美国科学院院士、美国华盛顿大学医学院Evan E. Eichler教授联合杰克逊基因组医学实验室Jan O. Korbel等研究团队,通过分析来自32个不同人类基因组的64个组装单倍型,完成了人类基因组和结构变异整合分析,对人类基因组的结构、变异和突变提供了基本的新见解。该项工作以“Haplotype-resolved diverse human genomes and integrated analysis of structural variation”为题发表在Science上。

研究团队对34个不相关的个体基因组进行从头测序,其中三个此前研究过的儿童样本进行了测序,完成了三个亲子三人组数据收集;并对两个样本的公开测序数据进行了分析。研究人员利用连续长读长(CLR)测序或高保真(HiFi)测序对所有基因组进行了测序。同时为了实现阶段化组装,为每个样本生成了相应的Strand-Seq数据,通过无参考组装方法成功生成了70个(64种无关)分阶段组装的人类单倍型。结果显示,阶段性基因组组装在碱基对水平上准确(QV>40),高度连续(重叠N50 > 25Mbp)且具有较低开关错误率。与CLR测序相比,HiFi产生的人类基因组组装的序列准确性更高。基于CLR测序,该研究组装的单倍型中有15个超过32 Mbp的重叠群N50,其中插入文库更大,序列覆盖率更高,是单分子实时(SMRT)细胞数量的一半(图1)。
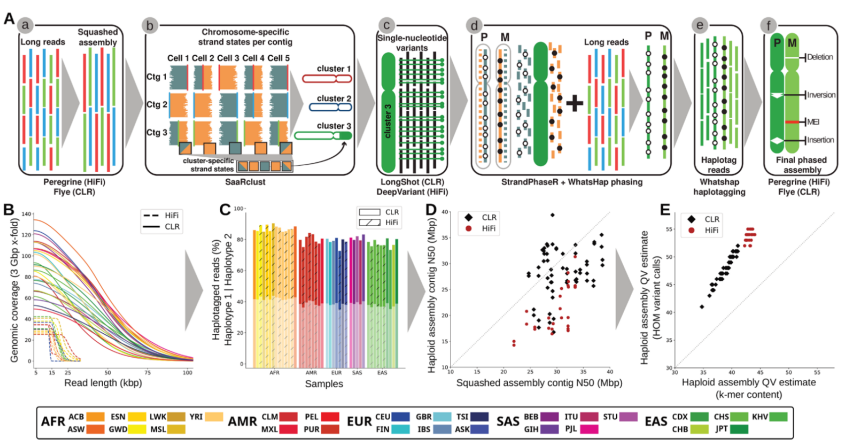
图1. 利用Strand-seq(PGAS)进行无三阶段性二倍体基因组组装。来源:Science
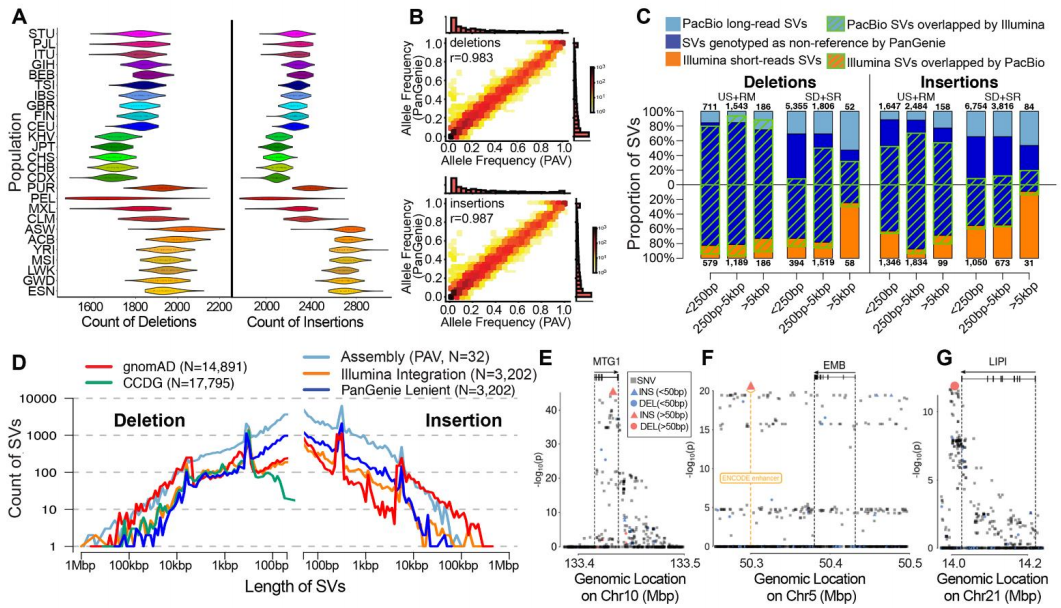
研究团队开发了阶段性组装变异体(PAV)调用器,以便对两个序列组装的单倍型与人类参考基因组GRCh38进行直接比较,从而发现遗传变异。结果显示,在每个二倍体人类基因组中平均可检测到24653个SVs、794,406个INDELs和3895,274个SNVs。为了将PAV调用与WGS发现的遗传变异进行比较,研究团队对来自1000GP的3202个样本进行了基于Illumina的短读长测序,结果显示平均77.4%的短读长测序组装检测到的SVs与长读长组装集是一致的,在短读长WGS调用集中只观察到29.6%的长读长SVs。在较小的SVs中,长读长测序组装的灵敏度较高,其中约83.3% (<250 bp)为新事件;短读长测序组装在大SVs(> 5 kbp)中表现出更高的灵敏度。最终,研究人员基于PAV确定了278个SV热点。此外,在64个单倍型中,共识别了106个非冗余位点,在映射到外显子内的不间断CTG或CGG重复插入的最大插入中,有5/7对应于与三联体重复不稳定疾病或脆性位点相关的基因,这些完美重复插入等位基因的发现为进一步研究三联体重复不稳定性提供了重要的参考。
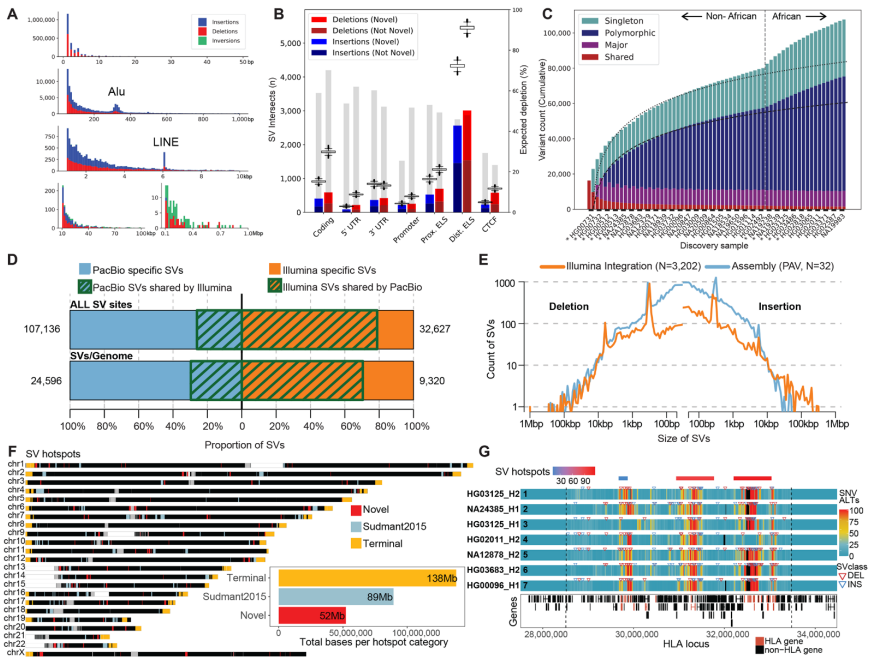
图2.变异体的发现和分布。来源:Science
根据分阶段的基因组组装,研究鉴定了一组完全序列解析的非参照MEI,包括7,738个Alus,1,175个LIHs和540个SVAs。研究发现,非参考SVA在VNTR拷贝数上比参考SVA显示出更大的变异性。SVA元件在其内部区域含有富含CpG的VNTR。
此外,研究发现每个样本平均有117个倒置。在30个样本中,通过光学图谱发现的72%的大片段插入和缺失(≥5 kbp)是完全序列解析的,与组装一致,但显示出比单个缺失事件更复杂的模式。研究人员预估,在人类群体中,每个阶段的基因组装中仍有大约35个长度为 > 50kbp的未解析区域,且存在5种或更多不同的SV单倍型。
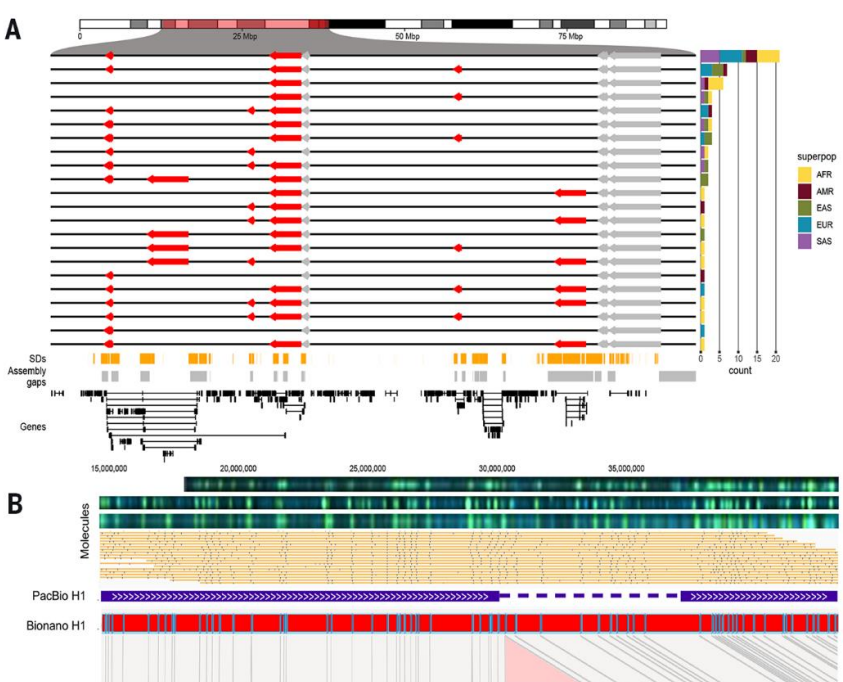
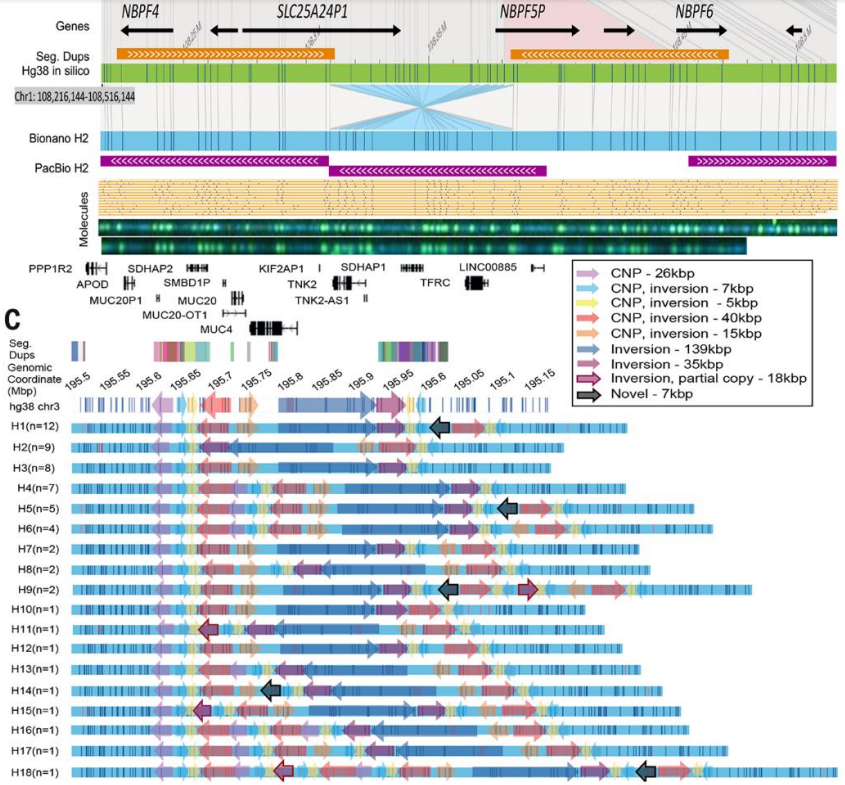
图3.结构变异的复杂模式。来源:Science
研究者使用15.5M SNVs、1.03M indels(1-49bp)和96.1k SVs的参考集来进行基因分型,并将这些变异分型到1000GP WGS数据集中观察预期的多样性模式。对长读长、短读长测序组装 SV以及PanGenie可进行基因分型的SV进行整合比较,结果显示,平均每个样本有167个大型CNVs(> 5 kbp),其中88.2%未被长读长测序组装集捕获。值得注意的是,研究发现42.5%(严格集)和59.9%(宽松集)的pangenies基因型SVs在短读长测序集中缺失。此外,该研究报告的SV-eQTL关联中有42%是新的。
单倍型相组装的可用性为在多个水平上探索基因组和SVs的祖先和群体遗传特性提供了支持。研究团队建立了Markov模型来识别包含祖先信息的SNVs,并根据Simons Genome Diversity Project (SGDP) 的群体遗传数据分配每个区块的祖先片段。结果显示这两种方法以及不同的测序平台产生高度一致的结果,且在家庭层面上,可以准确地分配父亲和母亲的单倍型,并将孩子的重组交换事件与父母的单倍型区分开来。在群体水平上,平均87.2%的组装序列可以被指定为祖先,来自非洲大陆的1000 GP样本显示出最大的一致祖先区域。
综上所述,该研究利用长读长测序技术和链特异性测序技术实现了高质量的单倍型解析人类基因组的从头组装,而无需亲子三人组数据,并发现了与基因表达、剪接和候选疾病位点有关的新关联。该研究对人类基因组的结构、变异和突变提供了基本的新见解,为今后对数千个人类基因组进行更系统的分析提供了框架。
 热文推荐
热文推荐

喜欢别忘了点“在看”呦!



 个人中心
个人中心
 我是园区
我是园区




