
近日,清华大学的研究团队表明,对来自肿瘤细胞的cfDNA占比进行分析,有助于癌症早筛和肿瘤定位。相关文章以“Tumor fractions deciphered from circulating cell-free DNA methylation for cancer early diagnosis”为题发表于期刊《Nature Communications》上。

为精准评估来自肿瘤细胞的cfDNA,量化其在总cfDNA中的占比,全基因组测序法(WGS)、ichorCNA法、ACE法近年来先后被提出,但这些方法各有各的劣势,无法解决根本上的难题,可靠稳健地评估来自肿瘤细胞的cfDNA占比。
清华大学研究团队注意到,在健康个体与肿瘤患者之间,DNA甲基化特征有着很大的差别,而近年来多项研究详细分析了两群体当中的差异甲基化位点/区域(DMPs/DMRs)。同时,cfDNA有多种来源,每一CpG位点的的甲基化水平本质上是造血细胞和其他组织的混合信号,包括肿瘤细胞。
于是,研究团队想到了通过cfDNA甲基化特征构建SRFD-Bayes模型,用去卷积的方法估计来自肿瘤细胞的cfDNA占比,并进一步对其定位。
注:去卷积法,是用于估计混合物(数据点)中每种细胞类型的比例以及每个细胞的基因表达水平(在同一数据点内)的算法。
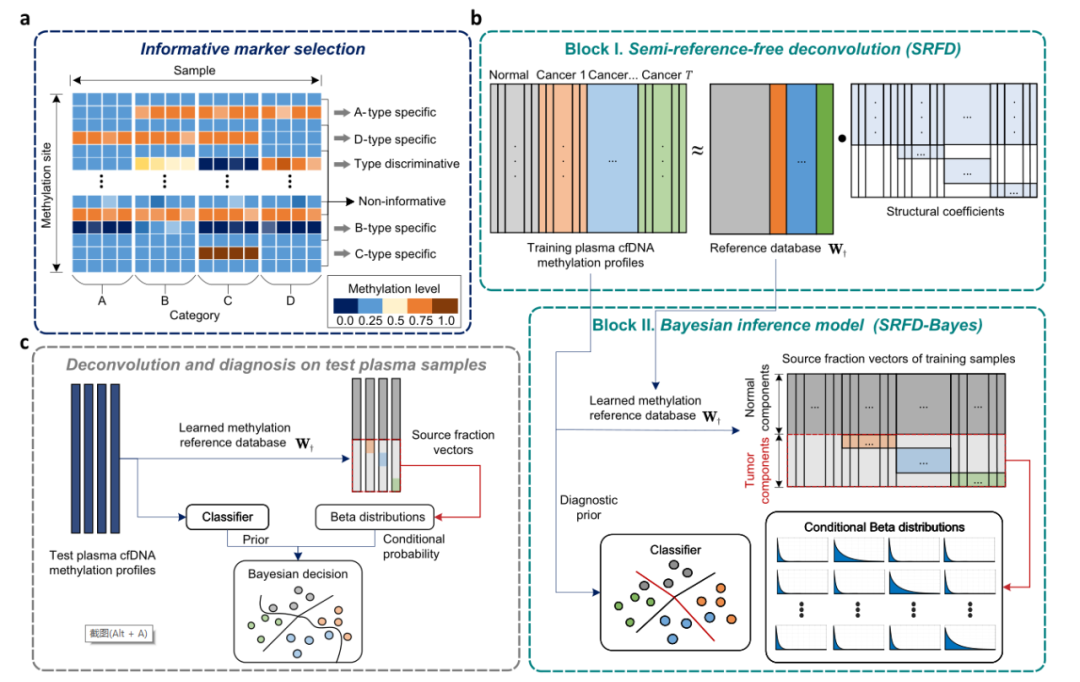
首先,研究团队基于矩阵范数设计了一个评分系统,以从大量的差异甲基化位点中识别类型判别(TD)和类型特异性(TS)甲基化标记。
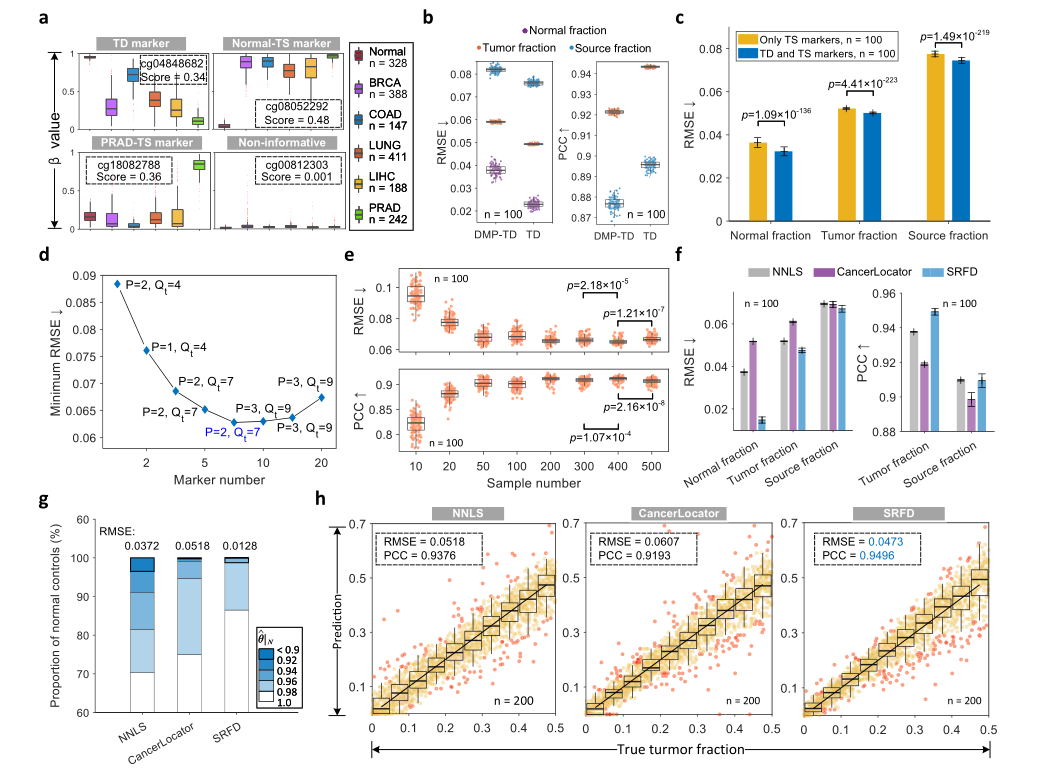
Informative marker selection and deconvolution results on simulations
接着,研究团队利用自由反卷积算法从混合血浆数据库中学习参考数据库。(包括656个正常血液DNA甲基化图谱和5组来源于癌症基因组图谱的肿瘤组织DNA甲基化数据,囊括浸润性乳腺癌、结直肠癌、肺癌、肝癌和前列腺癌五大癌种)
最后,研究团队将训练好的样本反卷积到单独的源分数向量中,每个癌种的数据都被当做是独立的Beta分布进行拟合,一同构建预诊断模型,也就是SRFD-Bayes模型。
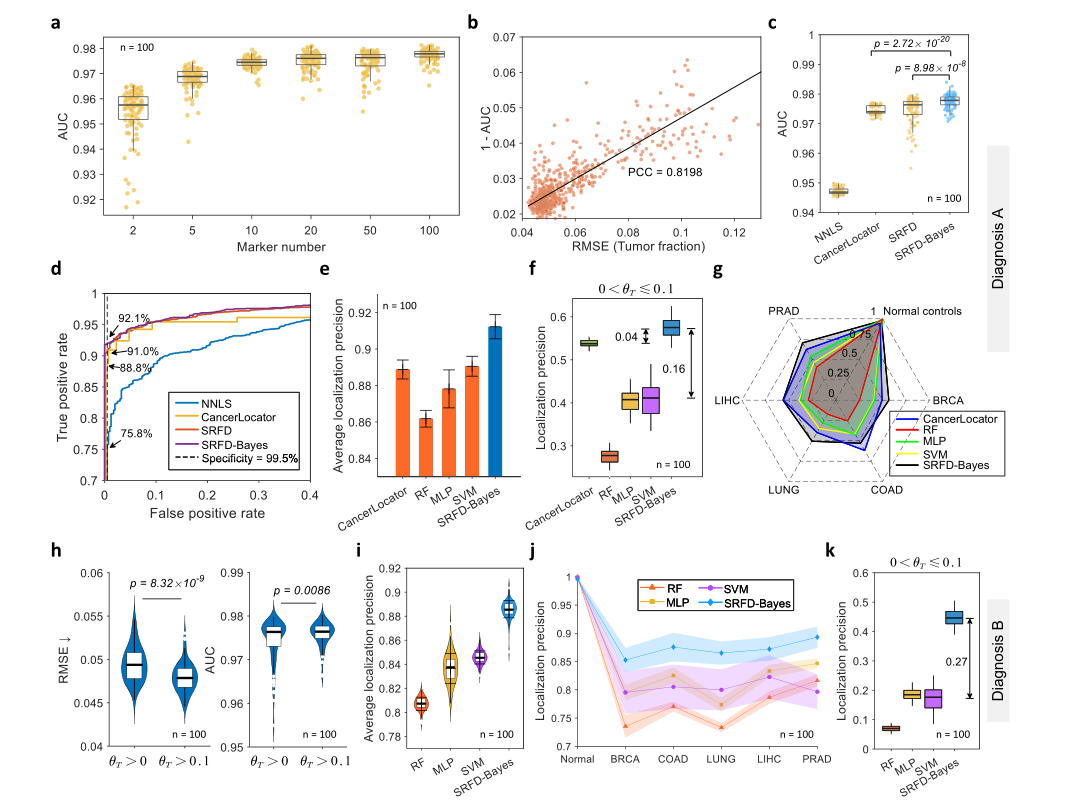
Diagnostic results on the simulation dataset
为在真实世界验证SRFD-Bayes模型的准确性,研究团队选取835名健康个体对照和1050名确诊的肝癌患者的数据进行测试,研究结果表明,SRFD-Bayes模型在癌症早期患者和健康个体上验证时,筛查早期癌症的灵敏度为86.1%,特异性为94.7%,肿瘤定位的平均准确性为76.9%,且来自肿瘤细胞的cfDNA占比,即SRFD-Bayes模型预测的肿瘤分数和肿瘤分期显著相关。
清华大学研究团队表示:“该模型基于晚期肿瘤样本的高度可迁移训练策略构建,在癌症早筛领域的价值巨大,可作为大规模肿瘤筛查或肿瘤进展监测工具。”
目前已有500+行业精英加入基因俱乐部
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()




 个人中心
个人中心
 我是园区
我是园区





