
责编 | 兮
细胞正常功能的维持离不开基因组DNA序列信息的精准传递——转录调控,转录则需要通过RNA聚合酶II(RNA polymerase II,RNAPII)的招募(recruitment)、起始(initiation)、暂停-释放(pausing-release)、延伸(elongation)等过程最终合成生物蛋白质大分子来执行下游功能 【1,2】。细胞命运则是由特定的转录程序来决定,且受到转录因子和表观遗传修饰(染色质调控因子)等在内的多层次因素的调节【3,4】。虽然过去几十年对于表观遗传调控的研究取得了巨大的成就,例如发现和鉴定了众多不同组蛋白、DNA或RNA修饰标志物的修饰酶(writer)、去修饰酶(eraser)和修饰识别蛋白(reader);但是大部分组蛋白的翻译后修饰(posttranslational modifications,PTMs)在染色质调节和转录过程中的作用主要是通过破坏PTMs相关的酶来间接推测出来的。
在众多的组蛋白修饰中,基因活化状态的标志物组蛋白H3上第4位的赖氨酸三甲基化(H3K4me3)是被研究得最为广泛的PTMs之一,在哺乳动物细胞中其可以被6种不同的COMPASS复合物催化生成【5-8】。由于H3K4me3在基因启动子附近的富集水平与基因表达水平正相关,因此长久以来H3K4me3一直被认为是可以促进基因转录起始的。这一假说可以被一些体外生化实验结果得以支持;此外,蛋白质谱数据也显示H3K4me3可以募集一些转录起始相关因子,例如TAF3和BPTF等【5,9】。但在细胞水平上,领域里却一直缺乏支持H3K4me3促进转录起始这一观点的直接有力证据(换句话说,H3K4me3在细胞内与基因转录调控的因果关系是不清楚的)。有意思的是,在酵母中敲除H3K4甲基转移酶Spp1可以使H3K4me3显著下降却不影响基因转录【10】,但COMPASS功能异常又会导致小鼠胚胎发育障碍和众多不同类型肿瘤的发生【11-15】。
那么,H3K4me3在细胞体内对基因转录到底有没有直接作用呢?2023年3月1日,美国纪念斯隆-凯瑟琳癌症中心Kristian Helin研究组在Nature上发表了题为:H3K4me3 regulates RNA polymerase II promoter-proximal pause-release的研究长文,对这一长期困扰学界的问题进行了回答。

在该研究中,研究人员发现H3K4me3在细胞体内是通过转录暂停-释放过程来调节基因表达,而不是以往认为的促进基因转录起始,并阐明了H3K4me3调控这一过程的全新机制。
由于传统的基于基因敲除或敲降等技术研究H3K4me3的方法很难排除次级影响,加上哺乳动物细胞中催化H3K4甲基化相关酶复合物之间存在补偿效应或非特异性的问题,导致在细胞体内很难对这其具体功能进行准确研究。因此研究人员首先建立了基于靶蛋白快速降解及可逆调控技术(degron)的COMPASS快速降解新系统(两个独立的系统:DPY30-mAID和RBBP5-FKBP,细胞系为小鼠胚胎干细胞(mESCs)),该新系统使得研究人员可以在不破坏细胞活力、细胞周期以及复合物内其他组成成分的情况下急性降解COMPASS成员达到研究H3K4甲基化的特异性功能的目的(图1)。
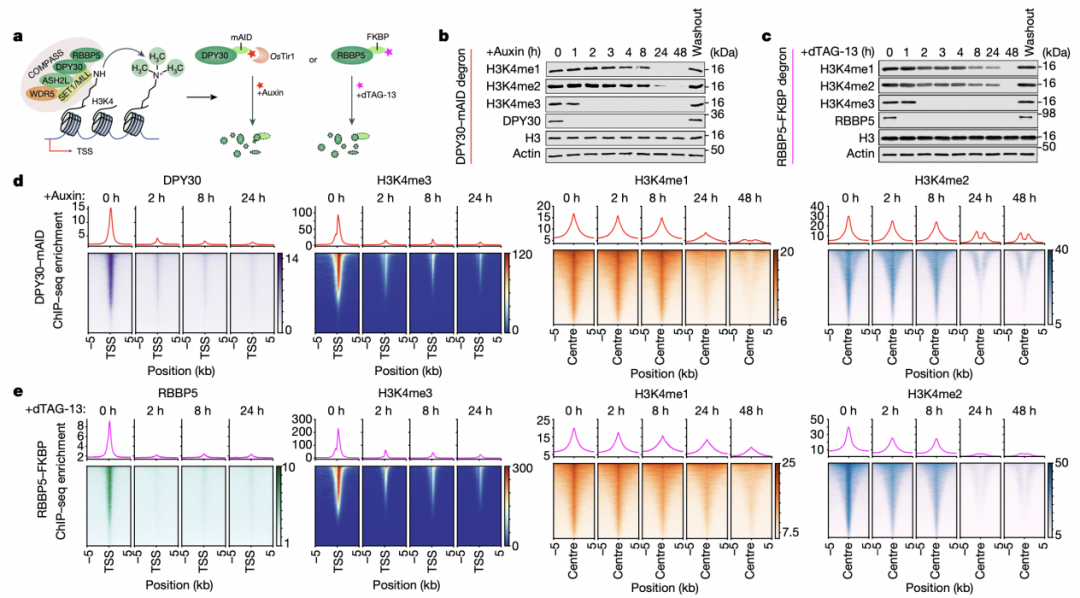
图1 H3K4me3的快速周转依赖去甲基化酶的主动去甲基化过程。
结果表明,在这两个降解系统中,随着诱导降解时间的延长,所有的H3K4甲基化(me1/2/3)可以被完全清除。有意思的是,相比H3K4me1和H3K4me2的完全丢失要等到24个小时的降解,H3K4me3的清除可以在惊人的2个小时内被完成。这一结果出人意料,因为一般认为组蛋白甲基化修饰的周转主要依靠细胞周期复制,至少需要10个小时以上。通过在DPY30-mAID系统中双敲除H3.3或H3K4me3去甲基化酶KDM5A/5B(dKO),研究人员发现H3K4me3的快速周转主要依赖去甲基化酶的主动去甲基化过程,这一新发现也拓展了对组蛋白修饰周转的认识。
随后,新生成熟RNA测序(SLAM-seq)结果则表明H3K4me3的快速丢失导致基因转录水平显著性降低。然而通过RNAPII的ChIP-seq和质谱,没有发现缺失H3K4me3后导致转录起始被抑制的证据,反而发现启动子附近RNAPII的富集明显增多,提示可能H3K4me3涉及转录暂停-释放过程。而且这些现象可以在KDM5 dKO细胞中被显著的延迟,说明这些影响确实是由于缺失H3K4me3本身导致的。与此一致的是,mNET-seq结果显示H3K4me3的缺失会导致RNAPII堆积在暂停位点上且其半衰期被显著延长;TT-seq数据表明RNAPII延伸速度也普遍下降。同时,mESCs的快速诱导分化(RA)实验也证明H3K4me3在RNAPII转录(从头)起始过程中不是必需的。
最后,通过基于APEX2技术的RNAPII临近蛋白生物素标记系统发现整合子(Integrator)复合体催化亚基INTS11在H3K4me3的这一转录调控过程中起着关键作用。基于这些新发现,研究人员也提出了新的H3K4me3调控基因转录的模型:快速周转的H3K4me3通过动态招募INTS11到基因启动子附近,INTS11则可以通过其核酸内切酶活性来促进处于暂停状态的RNAPII跨过+1核小体进行转录延伸。这些结果将进一步促进领域里对PTMs在基因转录过程中发挥的特异性作用机制的理解,也为研究H3K4me3相关调控因子在细胞命运决定和肿瘤发生发展中的具体作用提供了借鉴。
美国纪念斯隆-凯瑟琳癌症中心王华博士(目前为Xuejun Jiang实验室的博士后副研究员)是本文独立第一作者,著名表观遗传学家Kristian Helin(现为伦敦癌症研究所(ICR)的首席执行官兼主席)是该研究的通讯作者。此外,该研究工作得到了美国纪念斯隆-凯瑟琳癌症中心Xuejun Jiang(姜学军)教授等合作者的大力支持。
王华博士将加盟北京大学国际癌症研究院,团队正在招收具有表观遗传学、肿瘤学、细胞死亡(铁死亡)、基因转录调控和生物信息学等生物相关背景的研究生、博士后和科研助理、技术员。有意者可准备申请材料(个人简历、既往主要研究内容等)进行投递,应聘理由请标明:所应聘职位+姓名。

原文链接:
https://doi.org/10.1038/s41586-023-05780-8
制版人:十一
参考文献
1. Chen, F.X., E.R. Smith, and A. Shilatifard, Nat Rev Mol Cell Biol, 2018. 19(7): p. 464-478.
2. Adelman, K. and J.T. Lis, Nat Rev Genet, 2012. 13(10): p. 720-31.
3. Piunti, A. and A. Shilatifard, Science, 2016. 352(6290): p. aad9780.
4. Flavahan, W.A., E. Gaskell, and B.E. Bernstein, Science, 2017. 357(6348).
5. Lauberth, S.M., et al., Cell, 2013. 152(5): p. 1021-36.
6. Shilatifard, A., Annu Rev Biochem, 2012. 81: p. 65-95.
7. Miller, T., et al., Proc Natl Acad Sci U S A, 2001. 98(23): p. 12902-7.
8. Qu, Q., et al., Cell, 2018. 174(5): p. 1117-1126 e12.
9. Vermeulen, M., et al., Cell, 2010. 142(6): p. 967-980.
10. Murray, S.C., et al., bioRxiv, 2019: p. 709014.
11. Clouaire, T., et al., Genes Dev, 2012. 26(15): p. 1714-28.
12. Ang, Y.S., et al., Cell, 2011. 145(2): p. 183-97.
13. Jiang, H., et al., Cell, 2011. 144(4): p. 513-25.
14. Denissov, S., et al., Development, 2014. 141(3): p. 526-537.
15. Dorighi, K.M., et al., Mol Cell, 2017. 66(4): p. 568-576 e4.




转载须知
【非原创文章】本文著作权归文章作者所有,欢迎个人转发分享,未经作者的允许禁止转载,作者拥有所有法定权利,违者必究。




 个人中心
个人中心
 我是园区
我是园区





