
责编 | 兮
卵子受精后,随着胚胎经历大规模的染色质重编程
【1-5】
,生命萌芽之“密码”便逐步开启。组蛋白H3第9个赖氨酸的三甲基化
(H3K9me3)
是基因组异染色质的主要标记,它与胚胎发育密切相关
【6-9】
。然而,迄今人们对异染色质在人类早期胚胎发育过程中的重编程规律及其功能还知之甚少。另外,人类早期胚胎发育与小鼠等哺乳动物有较大差异,但是尚不清楚造成种间差异的因素是什么。
2022年7月7日,中山大学中山医学院王继厂课题组与孙逸仙纪念医院王文军教授团队合作在Cell Stem Cell上发表了题为
Dynamic reprogramming of H3K9me3 at hominoid-specific retrotransposons during human preimplantation development
的论文,
阐明了H3K9me3依赖性异染色质重塑在人类早期胚胎发育中的功能及其机制
,初步回答了上述问题。值得一提的是同济大学生命科学与技术学院高绍荣/刘晓雨/王晨飞团队与广东省第二人民医院欧湘红团队合作在Cell Stem Cell杂志上发表了类似的工作,
题为
Stage-specific H3K9me3 occupancy ensures retrotransposon silencing in human preimplantation embryos
。
该研究利用CUT&RUN技术,描绘了人类着床前胚胎发育过程中组蛋白H3K9me3修饰的动态变化图谱,并揭示了时期特异性H3K9me3修饰在逆转座子上的建立及其功能,发现了可能介导H3K9me3修饰建立的关键调控因子
(详见BioArt今日单独推送)
。
为了克服传统ChIP-seq细胞需求量大、而人类早期胚胎获取不易的困境,作者开发了新的微量细胞ChIP-seq技术
(AULiChIP-seq)
技术,将起始细胞量从之前的几千个细胞降到了50-100个细胞。随后,他们利用AULiChIP-seq技术绘制出首张人类植入前胚胎的H3K9me3修饰动态“景观图”:H3K9me3的分布具有空间特异性,其主要分布于基因间区域的内源性逆转录病毒
(ERV)
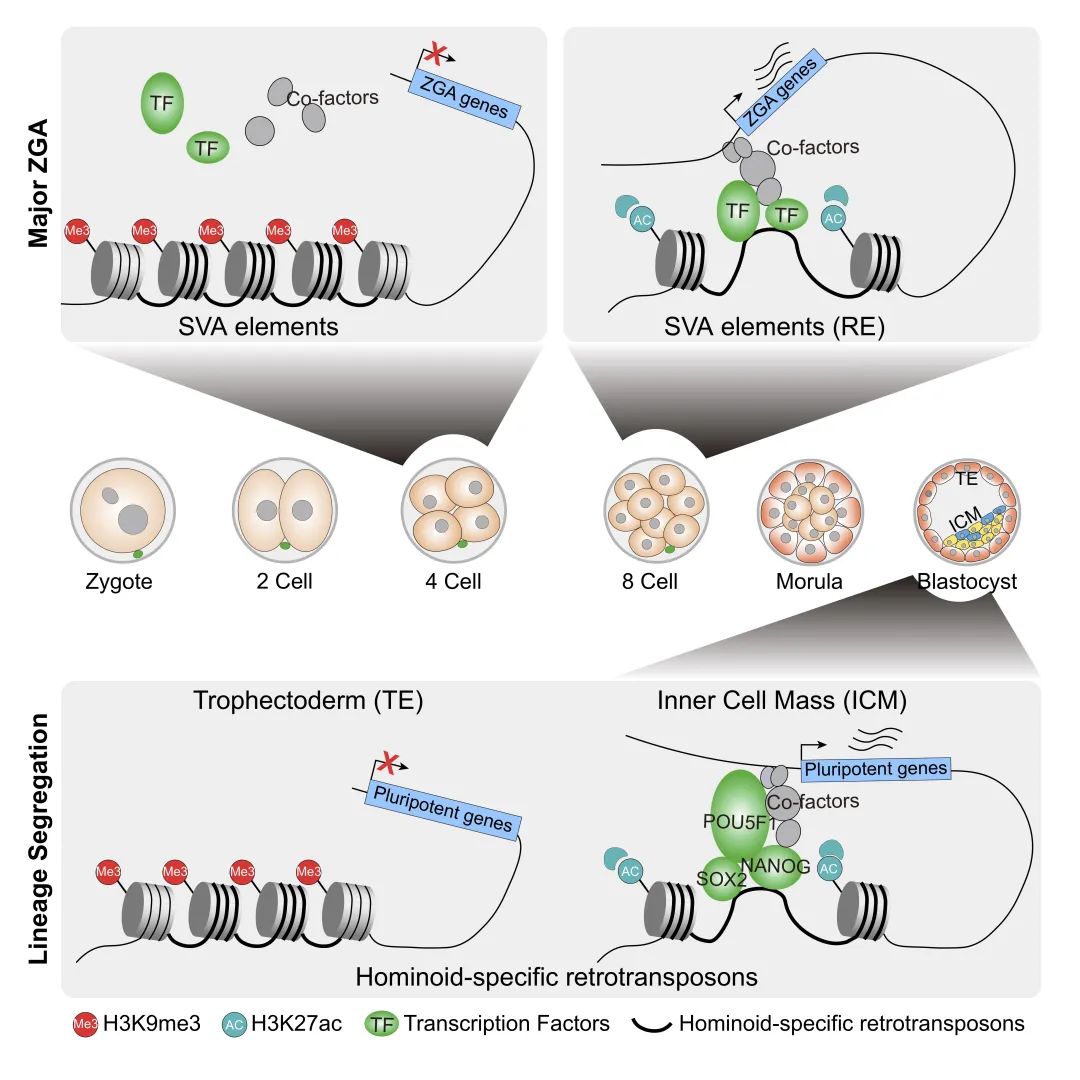
、SVA、LINE-1等反转录转座子上;另一方面,H3K9me3还呈现出时间特异性分布,其在胚胎发育的特定时期富集于细胞命运决定和谱系发育等相关基因周围,从而抑制这些基因在人类早期胚胎中的表达,确保对发育时序的精确调控。
与之前的研究结果一致
【2,3】
,作者发现,在小鼠和人类major ZGA启动前的胚胎中,大量major ZGA基因的启动子和转录起始位点已经处于染色质开放的状态。在小鼠二细胞晚期,伴随着ZGA基因体上的H3K9me3重编程,这些ZGA基因激活并大量表达。然而,与小鼠不同的是,人类major ZGA基因体上并不富集H3K9me3等抑制性组蛋白修饰,那么人类major ZGA为什么反而迟至4-8细胞胚胎时期,明显晚于小鼠呢?作者猜测这可能与增强子激活的时期有关。鉴于H3K9me3的时空特异性,他们发现,直至人类8细胞胚胎时期, SVA等类人猿特有的反转录转座子
(以下简称为类人猿反转座子)
才发生大规模的H3K9me3重编程而成为活化的增强子,从而促进major ZGA基因的表达。
为了进一步验证SVA区域的H3K9me3重编程对人类合子基因组激活的重要性,作者建立了针对反转座子的表观基因组编辑系统,结合其开发的适用于早期胚胎的微量细胞多组学分析技术Pathul-seq,在特异性地抑制人类早期胚胎SVA区域的H3K9me3重编程后,进行转录组和表观组分析。结果显示,在表观基因组编辑的胚胎中,SVA上H3K9me3修饰水平显著升高,其潜在调控的ZGA基因表达随之受到抑制,并表现出发育迟缓,囊胚发育率降低等表型,提示SVA抑制对人类早期胚胎发育有阻碍作用。作者还发现,在卵裂球部分性发育停滞的IVF胚胎中,SVA转录活性明显下降,伴随着部分ZGA基因的表达水平降低。这些结果表明,某些类人猿反转座子区域的H3K9me3重编程对于人类合子基因组激活与囊胚形成是不可或缺的。然而,出乎意料的是,进化年龄最年轻且具有转座能力的另一部分SVA和L1Hs
(一类人类特有的LINE-1
)却在整个人类早期胚胎发育时期保持H3K9me3和H3K27me3同时富集的状态
(即H3K9/K27me3 bivalency)
,这导致它们的转录活性被显著抑制,从而限制其在基因组上的“跳跃”。具有转座能力的反转座子是如何“抵抗”H3K9me3重编程的,值得今后进一步研究。
为了探讨H3K9me3依赖性异染色质重塑是否参与人类早期胚胎的其他重要发育过程如胚胎细胞谱系分离,作者整合多组学数据,对桑葚胚向囊胚的内细胞团
(ICM)
与滋养层细胞
(TE)
发育过程中H3K9me3的动态变化进行了分析。结果显示,TE特异性获得的H3K9me3 domain亦主要分布于类人猿反转座子
(如LTR12C)
上,且这些区域在ICM中处于开放状态。作者进一步证实,TE特异性获得的H3K9me3富集于类人猿反转座子上,阻碍多能性转录因子的结合,从而避免这些具有增强子活性的反转座子促进ICM特异性基因在TE中异位表达。因此,类人猿反转座子区域的H3K9me3 domain可能在TE“身份”的形成与维持上发挥一定作用。
最后,作者还比较了ICM与人类多能干细胞在表观基因组修饰上的异同,发现在ICM的某些反转座子上同时有H3K4me3和H3K9me3富集
(即H3K4/K9me3 bivalency)
,而这种独特的组蛋白双价修饰模式在5iLA等“原始态”人类多能干细胞中并不明显。特别地,H3K9me3在某些SVA位点的富集可促进DNA修复基因的表达,提示这些区域的H3K9me3富集对于维持人类多能性细胞基因组稳定性是至关重要的。上述发现为进一步提高人类“原始态”多能干细胞的质量提供了新的思路。
类人猿反转座子区域的H3K9me3依赖性异染色质重塑,促进人类早期胚胎发育
综上所述,
该研究揭示了哺乳动物早期胚胎发育中表观遗传调控快速进化的特点和规律,证实某些原本会破坏基因组稳定性的类人猿反转座子已进化成为对人类有益的重要调控元件,它们重塑了人类早期胚胎发育过程中的转录调控网络。该研究对优化IVF胚胎培养条件、提高试管婴儿发育率等临床实践,也具有重要的指导意义。
中山大学中山医学院博士生余汉文、硕士生陈漫琪、原副研究员胡远浪博士,和孙逸仙纪念医院区颂邦、李瑞岐博士为论文并列第一作者;王继厂教授、王文军教授和李瑞岐博士为论文共同通讯作者。该研究得到了南开大学刘林教授、英国皇家学会和皇家医学科学院院士Laurence D. Hurst教授等的大力支持。
值得一提的是,来自耶鲁大学的Andrew Zhuo Xiao对上述工作进行了评述。
附:王继厂教授课题组聚焦于人-动物异种嵌合研究,目标是在大动物体内培育出功能性人体器官,从而解决移植器官短缺这一难题。经过4年的积累,课题组初具研究特色和优势,近期在Cell Stem Cell、Cell Reports等国际知名期刊发表多篇学术论文。
王继厂教授课题组主要围绕以下三个方向开展研究:
(1)合成胚胎学:利用全能干细胞组装成类胚胎,在实验室中模拟和重现人类生命的产生和发展过程,破译人类早期胚胎发育的“密码”,目标是为复发性流产和出生缺陷等发育性疾病的临床诊治提供理论依据和干预策略;
(2)异种嵌合:开发和优化高效的人-动物异种嵌合体技术,进行人体器官发育的基础研究,目标是在动物(例如猪)体内培育功能性人体器官。
(3)灵长类脑发育:利用课题组独特的干细胞资源和异种嵌合技术,主要以脑类器官为模型,探讨包括人类在内的灵长类动物大脑发育和进化的分子机制,目标是为人类发育性脑病的临床诊治提供理论依据和干预策略。
目前,多项课题已取得一些突破性进展,急需新的力量加入,以加快推进相关研究。欢迎对上述研究方向有浓厚兴趣的志同道合者加入其课题组(可申请岗位有助理教授、博士后和研究生)。
https://jinshuju.net/f/ZqXwZt
或扫描二维码
投递简历
https://doi.org/10.1016/j.stem.2022.06.006
制版人:十一
1. Lu, F., Liu, Y., Inoue, A., Suzuki, T., Zhao, K., Zhang, Y. (2016) Establishing Chromatin Regulatory Landscape during Mouse Preimplantation Development. Cell 165, 1375-1388.
2. Wu, J., Huang, B., Chen, H., Yin, Q., Liu, Y., Xiang, Y., Zhang, B., Liu, B., Wang, Q., Xia, W., et al. (2016). The landscape of accessible chromatin in mammalian preimplantation embryos. Nature 534, 652-657.
3. Wu, J., Xu, J., Liu, B., Yao, G., Wang, P., Lin, Z., Huang, B., Wang, X., Li, T., Shi, S., et al. (2018). Chromatin analysis in human early development reveals epigenetic transition during ZGA. Nature 557, 256–260.
4. Xia, W., Xu, J., Yu, G., Yao, G., Xu, K., Ma, X., Zhang, N., Liu, B., Li, T., Lin, Z., et al. (2019). Resetting histone modifications during human parental-to-zygotic transition. Science 365, 353-360.
5. Borsos, M., Perricone, S.M., Schauer, T., Pontabry, J., de Luca, K.L., de Vries, S.S., Ruiz-Morales, E.R., Torres-Padilla, M.-E., and Kind, J. (2019). Genome–lamina interactions are established de novo in the early mouse embryo. Nature 569, 729-733.
6. Wang, C., Liu, X., Gao, Y., Yang, L., Li, C., Liu, W., Chen, C., Kou, X., Zhao, Y., Chen, J., et al. (2018). Reprogramming of H3K9me3-dependent heterochromatin during mammalian embryo development. Nat. Cell Biol. 20, 620–631.
7. Dodge, J.E., Kang, Y.K., Beppu, H., Lei, H., and Li, E. (2004). Histone H3-K9 methyltransferase ESET is essential for early development. Mol Cell Biol 24, 2478-2486.
8. Tachibana, M., Sugimoto, K., Nozaki, M., Ueda, J., Ohta, T., Ohki, M., Fukuda, M., Takeda, N., Niida, H., Kato, H., et al. (2002). G9a histone methyltransferase plays a dominant role in euchromatic histone H3 lysine 9 methylation and is essential for early embryogenesis. Genes Dev 16, 1779-1791.
9. Ninova, M., Fejes Toth, K., and Aravin, A.A. (2019). The control of gene expression and cell identity by H3K9 trimethylation. Development 146.
【非原创文章】本文著作权归文章作者所有,欢迎个人转发分享,未经作者的允许禁止转载,作者拥有所有法定权利,违者必究。








 个人中心
个人中心
 我是园区
我是园区




