Genome Biol l 陈子江/刘洪彬/黄涛/刘江合作揭示减数分裂同源重组表观调控新机制
收藏
关键词:
合作新机制揭示
资讯来源:BioArt + 订阅账号
发布时间:
2022-09-09

减数分裂是有性生殖生物体产生单倍体配子的特殊分裂方式,减数分裂的有序进行:一方面保证了遗传物质在生物世代间的稳定传递,另一方面通过同源重组产生等位基因的新组合,增加了遗传多样性
【1】
。减数分裂的分子调控机制研究一直是生命科学领域研究的焦点。减数分裂与生殖健康密切相关,其异常将导致生物体无法产生功能性配子或配子质量低下,最终引起不孕不育、自然流产或先天出生缺陷等
【2】
。
2022年9月6日,山东大学陈子江院士研究团队和与中科院基因组研究所刘江研究员团队通力合作,在以往工作基础上,共同在Genome Biology杂志上在线发表了文章
The histone modification reader ZCWPW1 promotes double-strand break repair by regulating cross-talk of histone modifications and chromatin accessibility at meiotic hotspots
,
揭示了组蛋白修饰识别分子ZCWPW1参与减数分裂同源重组调控的分子机制
,该研究
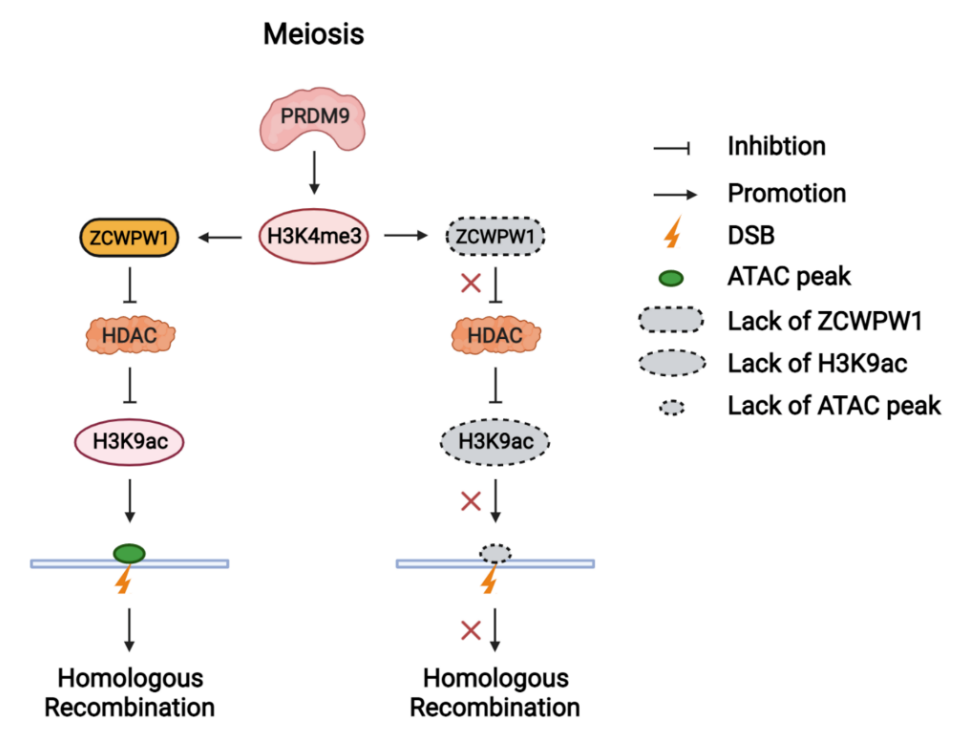
发现减数分裂重组热点处H3K9ac和染色质开放性的建立和维持依赖于H3K4me3催化酶PRDM9及其识别分子ZCWPW1,组蛋白甲基化阅读器ZCWPW1通过拮抗HDAC蛋白的去乙酰化活性维持H3K9ac,并进一步促进和维持重组热点处的染色质开放状态,从而为减数分裂DNA双链断裂同源重组修复提供保障。
 山东大学陈子江院士研究团队刘洪彬教授、黄涛副研究员长期关注减数分裂分子调控的机制研究。团队前期与香港大学深圳医院刘奎教授合作鉴定到一个减数分裂联会复合体中轴的稳固分子SCRE,发现SCRE蛋白通过结合联会复合体中轴组分SYCP1和SYCE3发挥稳定联会复合体的功能
(Nucleic Acids Res. 2019, 47(11):5670-5683)
;鉴定了一个新的减数分裂重组桥成分MEIOK21,证实MEIOK21通过与重组因子HSF2BP互作招募重组酶RAD51和DMC1到减数分裂DNA双链断裂
(Double strand break,DSB)
位点,并促进减数分裂同源重组的顺利进行
(Nucleic Acids Res. 2020, 48(12):6624-6639)
;发现组蛋白修饰识别分子Zcwpw1敲除导致雄鼠不育,精母细胞减数分裂阻滞在偶线期阶段,并伴有DSB修复异常和同源重组缺陷
(Sci Adv. 2019, 5(8):eaax1101)
;进一步利用多个减数分裂相关基因敲除小鼠模型和点突变小鼠模型,证实ZCWPW1特异性识别PRDM9催化的重组热点附近的H3K4me3和H3K36me3,并参与减数分裂DSB修复
(eLife. 2020, 9:e53459)
。然而,组蛋白修饰识别分子ZCWPW1参与减数分裂同源重组调控的分子机制仍不明确。
染色体上同源重组的数目和分布受到时间和空间水平的精细调控,减数分裂同源重组由程序性DSB形成起始,而DSB形成的位置并非随机分布,而是优先发生在重组热点处
【3】
。早期研究发现Prdm9为定义哺乳动物重组热点位置的关键基因
【4-6】
,尽管多项研究表明PRDM9介导的H3K4me3和H3K36me3控制着DSB的形成
【7】
,但目前对于任何可能读取这些遗传标记并因此调控DSB形成和减数分裂同源重组过程的蛋白知之甚少。除了组蛋白甲基化修饰,组蛋白乙酰化修饰
(H3K9ac)
也在重组热点附近显著富集,且两种组蛋白修饰存在一定的Crosstalk【8, 9】,然而调控重组热点处H3K4me3与H3K9ac Crosstalk的分子机制以及组蛋白修饰之间的Crosstalk如何协同调控减数分裂同源重组仍然未知。
在本研究中,团队利用多种基因敲除小鼠动物模型证实重组热点处H3K4me3和H3K9ac的建立以及ZCWPW1识别重组热点处的H3K4me3不依赖于DSB的产生,且ZCWPW1缺失并不影响重组热点处H3K4me3的建立。进一步研究发现,重组热点处H3K9ac和染色质开放性的建立和维持依赖于H3K4me3催化酶PRDM9及H3K4me3识别分子ZCWPW1。
研究人员进一步通过一系列分子、细胞和生化实验证实,ZCWPW1与多个去乙酰化酶和染色质重塑分子存在相互作用,ZCWPW1通过拮抗HDAC蛋白的去乙酰化酶活性维持重组热点处的H3K9ac水平,并进一步促进和维持重组热点处的染色质开放状态,最终促进减数分裂DSB的同源重组修复。在体细胞中异源表达生殖细胞特异分子ZCWPW1,发现ZCWPW1可以识别基因组中部分H3K4me3区域,且ZCWPW1结合区域的H3K9ac信号明显增强。同时,体细胞中异源表达ZCWPW1显著提高了体细胞的DSB修复能力和同源重组效率。
山东大学陈子江院士研究团队刘洪彬教授、黄涛副研究员长期关注减数分裂分子调控的机制研究。团队前期与香港大学深圳医院刘奎教授合作鉴定到一个减数分裂联会复合体中轴的稳固分子SCRE,发现SCRE蛋白通过结合联会复合体中轴组分SYCP1和SYCE3发挥稳定联会复合体的功能
(Nucleic Acids Res. 2019, 47(11):5670-5683)
;鉴定了一个新的减数分裂重组桥成分MEIOK21,证实MEIOK21通过与重组因子HSF2BP互作招募重组酶RAD51和DMC1到减数分裂DNA双链断裂
(Double strand break,DSB)
位点,并促进减数分裂同源重组的顺利进行
(Nucleic Acids Res. 2020, 48(12):6624-6639)
;发现组蛋白修饰识别分子Zcwpw1敲除导致雄鼠不育,精母细胞减数分裂阻滞在偶线期阶段,并伴有DSB修复异常和同源重组缺陷
(Sci Adv. 2019, 5(8):eaax1101)
;进一步利用多个减数分裂相关基因敲除小鼠模型和点突变小鼠模型,证实ZCWPW1特异性识别PRDM9催化的重组热点附近的H3K4me3和H3K36me3,并参与减数分裂DSB修复
(eLife. 2020, 9:e53459)
。然而,组蛋白修饰识别分子ZCWPW1参与减数分裂同源重组调控的分子机制仍不明确。
染色体上同源重组的数目和分布受到时间和空间水平的精细调控,减数分裂同源重组由程序性DSB形成起始,而DSB形成的位置并非随机分布,而是优先发生在重组热点处
【3】
。早期研究发现Prdm9为定义哺乳动物重组热点位置的关键基因
【4-6】
,尽管多项研究表明PRDM9介导的H3K4me3和H3K36me3控制着DSB的形成
【7】
,但目前对于任何可能读取这些遗传标记并因此调控DSB形成和减数分裂同源重组过程的蛋白知之甚少。除了组蛋白甲基化修饰,组蛋白乙酰化修饰
(H3K9ac)
也在重组热点附近显著富集,且两种组蛋白修饰存在一定的Crosstalk【8, 9】,然而调控重组热点处H3K4me3与H3K9ac Crosstalk的分子机制以及组蛋白修饰之间的Crosstalk如何协同调控减数分裂同源重组仍然未知。
在本研究中,团队利用多种基因敲除小鼠动物模型证实重组热点处H3K4me3和H3K9ac的建立以及ZCWPW1识别重组热点处的H3K4me3不依赖于DSB的产生,且ZCWPW1缺失并不影响重组热点处H3K4me3的建立。进一步研究发现,重组热点处H3K9ac和染色质开放性的建立和维持依赖于H3K4me3催化酶PRDM9及H3K4me3识别分子ZCWPW1。
研究人员进一步通过一系列分子、细胞和生化实验证实,ZCWPW1与多个去乙酰化酶和染色质重塑分子存在相互作用,ZCWPW1通过拮抗HDAC蛋白的去乙酰化酶活性维持重组热点处的H3K9ac水平,并进一步促进和维持重组热点处的染色质开放状态,最终促进减数分裂DSB的同源重组修复。在体细胞中异源表达生殖细胞特异分子ZCWPW1,发现ZCWPW1可以识别基因组中部分H3K4me3区域,且ZCWPW1结合区域的H3K9ac信号明显增强。同时,体细胞中异源表达ZCWPW1显著提高了体细胞的DSB修复能力和同源重组效率。
综上所述,
本研究阐释了组蛋白修饰相关分子(Writer、Reader)协同调控减数分裂同源重组热点处组蛋白修饰和染色质开放性的分子机制,同时发现体细胞中异源表达生殖细胞特异分子ZCWPW1显著提高DSB修复能力。以上新发现拓展了减数分裂表观遗传调控的新视角,丰富了同源重组修复的基础理论。
山东大学陈子江院士,刘洪彬教授,黄涛副研究员和中科院基因组研究所刘江研究员为该论文的共同通讯作者。山东大学生殖医学研究中心黄涛博士、包子游硕士、王诗语博士和吴心悦博士、中科院基因组研究所袁慎立博士
(现哈佛医学院博士后)
为该论文的共同第一作者。该工作还得到了香港中文大学路钢教授、山东大学高等医学研究院刘招舰教授、浙江大学黄俊教授和武汉大学陈强教授的大力支持。目前陈子江院士团队欢迎对生殖与发育、男科学、妇幼健康、遗传学、生物信息、环境与健康、医学人工智能、医用物理及材料学等方向感兴趣的青年才俊加盟
(http://www.sduivf.sdu.edu.cn/info/1031/1289.htm)
。
原文链接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-022-02758-z
1. https://pubmed.ncbi.nlm.nih.gov/32374261/
2. https://pubmed.ncbi.nlm.nih.gov/31453335/
1. Hunter N: Meiotic Recombination: The Essence of Heredity. Cold Spring Harb Perspect Biol 2015, 7.
2. Biswas L, Tyc K, El Yakoubi W, Morgan K, Xing J, Schindler K: Meiosis interrupted: the genetics of female infertility via meiotic failure. Reproduction 2021, 161:R13-R35.
3. Smagulova F, Gregoretti IV, Brick K, Khil P, Camerini-Otero RD, Petukhova GV: Genome-wide analysis reveals novel molecular features of mouse recombination hotspots. Nature 2011, 472:375-378.
4. Baudat F, Buard J, Grey C, Fledel-Alon A, Ober C, Przeworski M, Coop G, de Massy B: PRDM9 is a major determinant of meiotic recombination hotspots in humans and mice. Science 2010, 327:836-840.
5. Myers S, Bowden R, Tumian A, Bontrop RE, Freeman C, MacFie TS, McVean G, Donnelly P: Drive against hotspot motifs in primates implicates the PRDM9 gene in meiotic recombination. Science 2010, 327:876-879.
6. Parvanov ED, Petkov PM, Paigen K: Prdm9 controls activation of mammalian recombination hotspots. Science 2010, 327:835.
7. Diagouraga B, Clement JAJ, Duret L, Kadlec J, de Massy B, Baudat F: PRDM9 Methyltransferase Activity Is Essential for Meiotic DNA Double-Strand Break Formation at Its Binding Sites. Mol Cell 2018, 69:853-865 e856.
8. Lam KG, Brick K, Cheng G, Pratto F, Camerini-Otero RD: Cell-type-specific genomics reveals histone modification dynamics in mammalian meiosis. Nat Commun 2019, 10:3821.
9. Spruce C, Dlamini S, Ananda G, Bronkema N, Tian H, Paigen K, Carter GW, Baker CL: HELLS and PRDM9 form a pioneer complex to open chromatin at meiotic recombination hot spots. Genes Dev 2020, 34:398-412.
【非原创文章】本文著作权归文章作者所有,欢迎个人转发分享,未经作者的允许禁止转载,作者拥有所有法定权利,违者必究。








 药选址
药选址





