
背景介绍

2017年,阅尔基因(NuProbe)美国创新中心核心成员,莱斯大学David Yu Zhang研究小组开发了一种等位基因特异性PCR富集技术——抑制探针置换扩增技术(Blocker Displacement Amplification,BDA)。BDA技术利用PCR扩增中引物、抑制探针和模板DNA结合的热力学差异,实现对野生型模板扩增的抑制,扩大了低频突变信号,从而大大提高了低频变异检测灵敏度。
2021年5月3日,该团队在Nature子刊Nature Biomedical Engineering(IF=19)上发表了题为“Selective multiplexed enrichment for the detection and quantitation of low-fraction DNA variants via low-depth sequencing”的学术论文。研究团队在BDA技术的基础上,推出了多重抑制探针置换扩增技术(multiplex blocker displacement amplification,mBDA)。mBDA技术仅需250X测序深度即可检测到VAF低至0.019%的低频基因变异。不同于其它突变等位基因富集技术,mBDA 可以很好地扩展到多重Panel(80重的扩增子的Panel可以实现300倍的中位富集倍数)。经过mBDA技术富集过后的文库,样本中低频变异等位基因的VAF得到放大,远远超过NGS系统误差,因此只需相对较低的测序深度,且不需要通过额外的错误校正,即可检测到低频变异,极大地提高了检测特异性;mBDA的富集效率在不同样本、不同文库之间极其稳定,可以以95%的准确度精确定量2倍差异的初始样本变异。mBDA-NGS技术大幅减少测序数据量(可减少100倍),可以让各种中低通量测序仪以较低的测序成本实现低频基因变异的检测。


mBDA检测技术的性能

图:mBDA-NGS法实验流程及低频突变检测性能。来源:Nature Biomedical Engineering
性能验证实验显示,使用mBDA-NGS法对混合细胞系进行检测,起始样本量为300ng基因组DNA,LoD最低可至0.019%;在变异序列比例方面(variant-read fraction ,VRF), 相比标准多重扩增子文库NGS测序法的0.2%,mBDA法可达到30%,其富集效率在150倍以上(中位数300倍);在中靶率基本相同的前提下(mBDA 81.4% vs. 标准扩增子法 89.6%),mBDA法极大减少了检测所需的测序数据量,从而降低测序成本;与探针捕获法相比,mBDA可将文库构建时间从24小时缩短至6小时。

mBDA-NGS 检测肿瘤低频变异
大部分qPCR、Sanger和NGS测序分析只能检测1%以上的VAF,因而不利于发现来自肿瘤亚克隆的低频突变。但这些突变可能在后续的靶向治疗选择压力下,发展成为耐药突变,导致治疗失败和疾病复发。因此,对低丰度突变位点的检测可以为患者制定个性化治疗方案提供信息,改善患者预后。
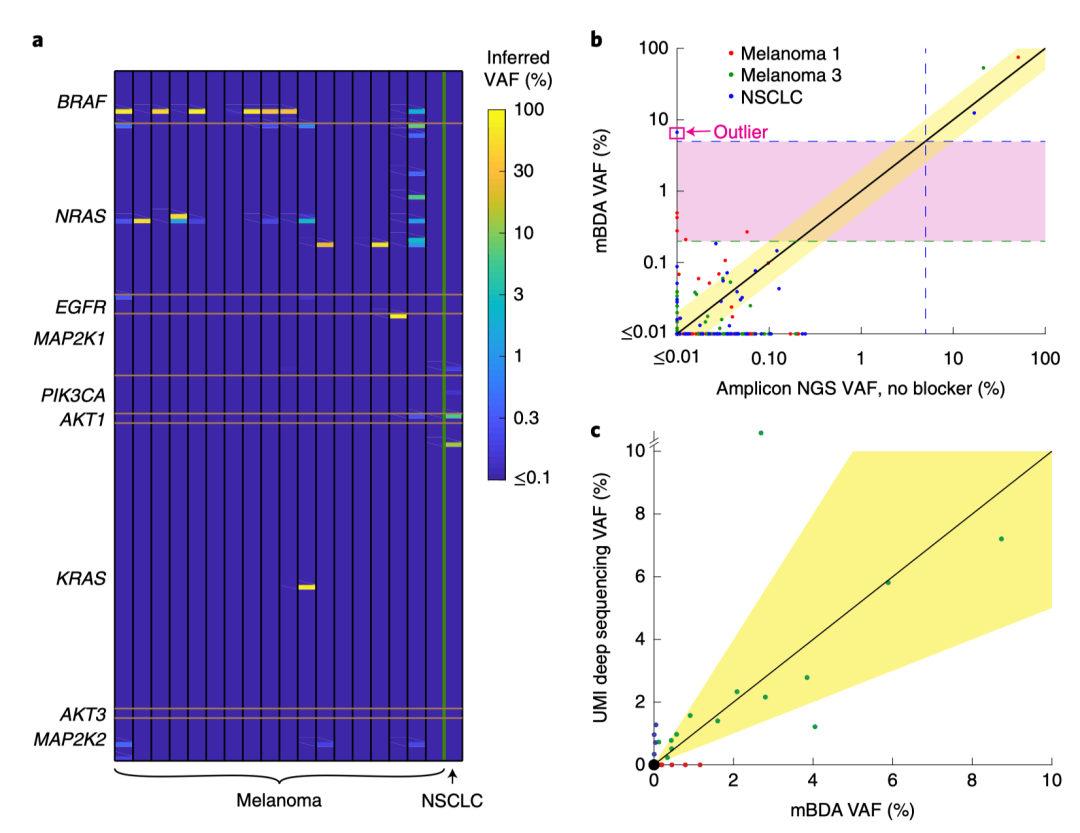
图:mBDA-NGS法用于肿瘤样本检测。来源:Nature Biomedical Engineering
针对肿瘤组织样本低频突变检测,研究人员参考COSMIC数据库,设计了一个16-plex、覆盖9个黑色素瘤相关基因、145个常见突变的mBDA-NGS panel,并对19例肿瘤冷冻样本(包括18例黑色素瘤和1例非小细胞肺癌,肿瘤细胞含量大于75%)进行检测。结果显示,所有样本均检测到主克隆变异(VAF≥5%),除此之外在7例样本中(37%)还检测到亚克隆突变(VAF在0.2%和5%之间),显示了肿瘤异质性的广泛存在,其亚克隆中的变异是导致靶向治疗耐药的机制之一。在传统的肿瘤组织样本检测中,由于大多数Panel测序深度小于10,000X,因此这样的低频变异会被遗漏。
mBDA-NGS技术也适用于cfDNA检测。在相同起始量情况下(6.8ng-18.9ng),研究人员对12例来自IV期非小细胞肺癌样本的cfDNA样本,同时使用mBDA NGS panel(14个基因共31个热点突变)和基于UMI的扩增子法深度测序分别进行检测。与基于UMI的深度测序相比,mBDA NGS阳性一致率为78%(14/18),阴性一致率为98.6%(503/510);在定量方面,大多数变异位点在2倍区间之内(0.5-2)。同时,相比于深度测序平均25,000X的测序深度,mBDA-NGS仅需250X测序深度,测序成本减少了近100倍。

mBDA用于评估细胞系污染
细胞系是重要的科研材料,但细胞污染在实验室里是普遍存在的。有证据表明,超过24%的现有细胞系或多或少都受到 HeLa 细胞系的污染。由此,很多知名期刊已经将细胞系鉴定证据作为发表前必要的提交材料。传统的检测方法如STR等常规的基于多态性的检测难以检测出微量或者未知来源的细胞污染。借助mBDA技术超高灵敏度的变异检测能力,结合NGS或qPCR可以进行更高灵敏度、高可靠性的细胞污染鉴定。
研究人员使用NA18537细胞系的gDNA作为野生型,按0.07~0.25%的比例混入不同来源的人gDNA,人工模拟微量细胞污染的样本。选取80个在NA18537中纯合的SNP位点构建80-plex mBDA NGS panel,结果表明,在检测起始量300ng的水平上,每个SNP仅需要约250X即可达到最低0.019%的VAF检测限。该研究中所有22个文库的测序可以在同一张Illumina MiSeq flowcell上完成。使用传统的扩增子建库法,一张flowcell仅能容纳两个文库,且有约20%的假阴性结果(VAFs > 0.02%)。通过mBDA技术对来自22个样本,共计1,760个位点检测,总体变异检出的特异性为98.42%,检出灵敏度在95.12%以上。这一数据已经达到甚至超过了大多数基于UMI的NGS方法,并且使用的reads数减少50倍。相比而言,常规扩增子测序按照同样的0.019%检测限,其检测特异性为100%,但灵敏度在纯合和杂合样本中分别只有69.23%和9.52%。
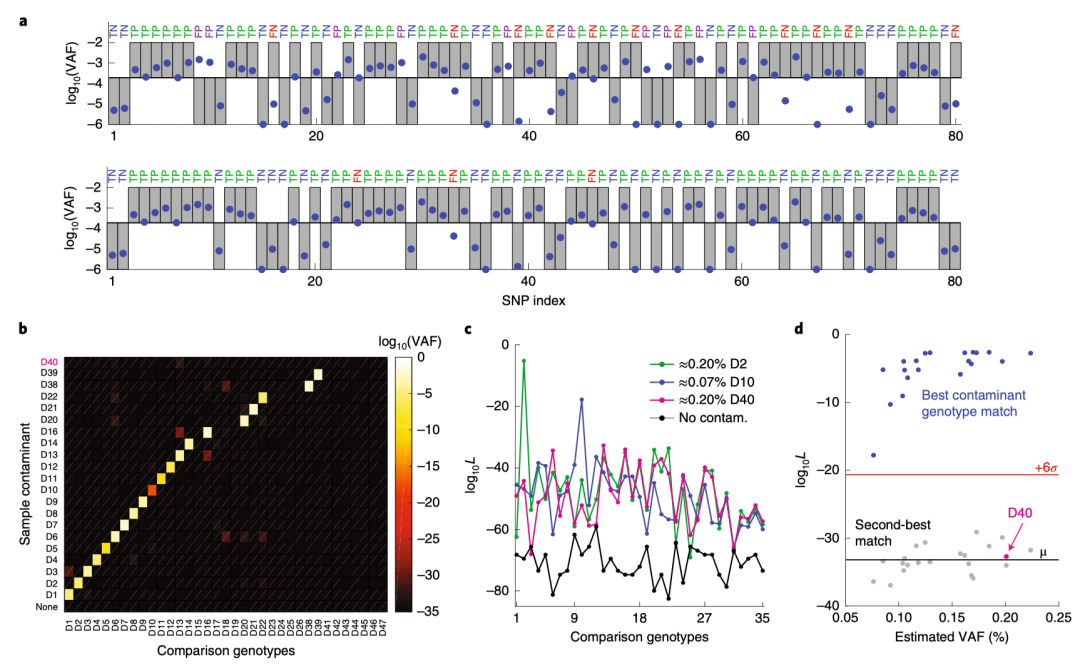
图:mBDA-NGS评估细胞系污染状况,来源:Nature Biomedical Engineering
随后,研究团队将这22个文库与35个(包含与前者重叠的21个)对照样本进行联合分析,并定义了似然值(likehood,\(L_{j}=\prod_{i=1}^{80} p_{i, j}\))来衡量两两之间变异的匹配程度。结果显示,所有实验样本中,基因型完全匹配的对照样本L值最大。对应基因型的组里,污染成分越少,对应的L值也越低,L值最低的D10对应很低的污染率(VAF=0.07%),但依然可以显著鉴别出来(\(P<10^{-9}\))。值得一提的是,实验组中的D40样本不在对照组中,L值在所有对照样本中都很低,但都显著高于无污染的gDNA样本。

总结

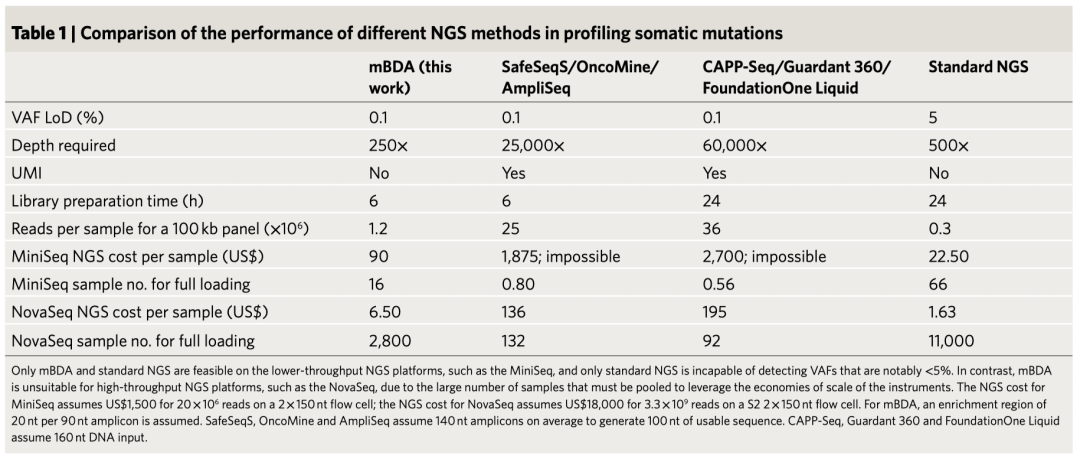
图:不同技术(NGS法)检测肿瘤体细胞变异性能比较。来源:Nature Biomedical Engineering
迄今为止,为了达到VAF 0.1%的检出限,所有的NGS panel都采用了UMI结合超深度测序(大于25,000X)的技术路径。这使得低频基因变异检测只能在高通量的NGS测序仪上进行,如NovaSeq,HiSeq X10等。然而这些高通量NGS测序仪更适合大型科研或服务项目,对大多数医院和参考实验室开展院内检测来说,在单次开机样本凑样、仪器运行时间、硬件成本等临床实际操作方面并不完全匹配,使得很多医院不得不外送至第三方大型实验室,给临床诊疗的实时性和合规性带来困扰,导致仍有大量癌症患者未能受益于NGS检测。通过mBDA-NGS技术,使用250X的测序深度即可对低频突变进行定量检测,从而使小型中低通量的测序仪器也能以较低的成本实现低频变异的检测。而小型中低通量NGS测序仪(如Miniseq,Nextseq,MGI200,Ion Torrent等)由于其价格适宜(例如约5万美元的MiniSeq)、凑样难度低、运行时间短,结合测序数据量降低减少了分析及数据存储成本,更适合临床需求。在肿瘤伴随诊断、术后监测、早筛早诊方面,mBDA技术均有助于促进更多的医院、实验室以去中心化的模式开展准确、快速、费用合理的检测,有望使更多的癌症患者接受基因检测,从而获得更有针对性的治疗。
· END ·
 热文推荐
热文推荐

喜欢别忘了点“在看”呦!



 个人中心
个人中心
 我是园区
我是园区




