
撰文 | 十一月
RNA修饰最早发现于20世纪50年代,如今已经有超过150种不同的RNA修饰被发掘和描述
【1-2】
。最常见的RNA修饰之一是m6A,该修饰常出现于DRACH
(D:A\G\U,R=A\G,H=A\C\U)
基序,影响RNA的结构、稳定性、剪接以及翻译
【3】
。在动物模型中,破坏m6A稳态影响干细胞的调节、生育能力和发育过程;而在人类中,这种修饰在癌症、细胞命运的转变和决定、发育和疾病中均起着重要作用
【4】
。但是目前为止所有的实验方法只能够提供RNA位点信息数据,但是对于单个RNA分子修饰状态的信息是缺失的。
2022年11月10日,新加坡国立大学Jonathan Göke研究组与泰国朱拉隆功大学Alexandre Thiery研究组合作在Nature Methods上发表了文章
Detection of m6A from direct RNA sequencing using a multiple instance learning framework
,
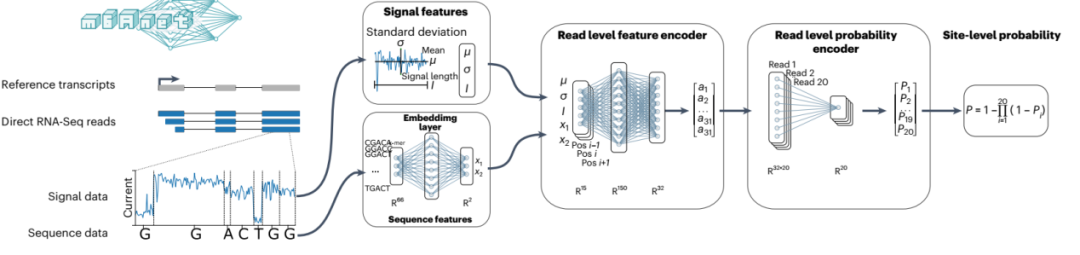
提出了一种基于神经网络的方法m6Anet,利用多实例学习框架处理数据中的缺失,对不同的细胞系和物种均具有较高的识别精度,可以从一次直接的RNA测序中捕获m6A的转录组范围内量化信息。
转录组范围内的RNA修饰实验的鉴定主要可以通过三种方法实现,其一是给予特异性结合修饰核苷酸抗体的免疫沉淀方法,比如MeRIP-Seq、m6A-Seq、PA-m6A-Seq、m6A-CLIP/IP、miCLIP、m6A-LAIC-Seq、m6ACE-Seq和m6A-Seq2等;其二是通过基于化合物选择性地与修饰的核糖核苷酸反应的化学方法,比如Pseudo-Seq3以及 AlkAniline-Seq;其三是通过使用特定的酶来选择性地区分修饰和未修饰的碱基,比如Mazter-Seq、m6A-REF-Seq或者DART-Seq等。这些方法的相似之处在于在诱导周围核苷酸发生变化后分离RNA,然后进行逆转录和短读cDNA测序来检测这些变化。虽然这些方法提供了RNA修饰位点的转录组范围图谱,但它们受到特异性抗体或化合物可用性的限制,而且这些测序结果并不包含单个RNA分子的信息。使用纳米孔进行RNA测序可以克服以上提到的这些限制,纳米孔利用RNA分子通过纳米孔隙时的电流强度推断RNA序列
【5】
。与未修饰的核苷酸相比,修饰过的核苷酸会发出不同的信号强度,这使得使用监督或比较方法可以计算出每个RNA分子的修饰位点。
为了训练出能够对单个RNA分子m6A修饰进行识别的工作模型,作者们从以单核苷酸分辨率识别m6A的m6ACE-Seq中获得的进行的数据训练。为了提取特征用于模型训练和预测,作者们将纳米孔原始信号分割到它们在转录组中的各自位置。由于m6A修饰发生在DRACH基序上,作者们从m6Anet的这些数据中删除了所有非DRACH基序。RNA-seq数据采用的是HCT116细胞系的结果,与之相匹配的m6ACE-Seq数据是新加坡纳米孔表达项目。
为了评估 m6Anet数据训练模型的表现,作者们对来自HEK293T细胞RNA-seq数据以及m6ACE-Seq数据、miCLIP数据进行测试。另外,作者们也将m6Anet数据训练模型与EpiNano、MINES、Tombo以及nanom6A的模型精确度进行对比。在所有的模型方法中,作者们发现m6Anet数据训练模型能够对备选m6A提供最准确的预测。
为了检测m6Anet数据训练模型能否从新的细胞系或者不同的物种m6A修饰进行预测,作者们使用了两个人类细胞系以及一个拟南芥的数据库。数据表明m6Anet可以稳健地推广到其他细胞系,而且不会因为细胞类型特定的数据而损失准确性。虽然特定物种的模型将提供最好的结果,但在缺乏特定物种的训练数据的情况下,即使使用默认的人类训练模型,m6Anet仍然可以提供准确的m6A修饰的预测。
为了评估特定技术m6A位点所预测的是否是有效的m6A位点,作者们确定了对m6A写入器METTL3缺失敏感的位置,通过使用野生型与METTL3-KO细胞系进行比较,绘制出HEK293T细胞系中的m6A位点。m6Anet模型的特异性预测可以对METTL3敲除表现出敏感性。
为了说明预测人类细胞系数据中每分子修饰的能力,作者们从HEK293T野生型和METTL3-KO细胞系中提取了候选m6A位置和概率,m6Anet可以捕获了潜在修饰率的变化,或用于比较一个RNA样本分子内的位点,或用于估计多个样本或不同条件下m6A丰度的位点特异性和全局差异。
总的来说,作者们通过神经网络训练建立了一个新的单分子RNA上m6A修饰的预测方法m6Anet,可以对不同细胞系以及不同物种中的m6A修饰进行高精度预测,并且可以达到转录组范围内m6A修饰概率的定量预测。
原文链接:
https://doi.org/10.1038/s41592-022-01666-1
制版人:十一
1. Boccaletto, P. et al. MODOMICS: a database of RNA modification pathways. 2017 update. Nucleic Acids Res. 46, D303–D307 (2018).
2. Dunin-Horkawicz, S. MODOMICS: a database of RNA modification pathways. Nucleic Acids Res. 34, D145–D149 (2006)
3. Wang, X. et al. N6-methyladenosine modulates messenger RNA translation efficiency. Cell 161, 1388–1399 (2015).
4. Jonkhout, N. et al. The RNA modification landscape in human disease. RNA 23, 1754–1769 (2017).
5. Garalde, D. R. et al. Highly parallel direct on an array of nanopores. Nat. Methods 15, 201–206 (2018).

【原创文章】BioArt原创文章,欢迎个人转发分享,未经允许禁止转载,所刊登的所有作品的著作权均为BioArt所拥有。BioArt保留所有法定权利,违者必究。











 个人中心
个人中心
 我是园区
我是园区





