植物育种利用减数分裂过程中同源染色体重组形成新的等位基因。
大麦(Hordeum vulgare)单花粉核基因分型可以在受精前测量配子的减数分裂重组率,而不需要分离群体。
由于花粉的DNA含量有限,现
在的方法依赖于每个花粉核的全基因组扩增(WGA),限制了分析样品的数量。
德国莱布尼茨植物遗传和作物植物研究所Leibniz Institute of Plant Genetics and Crop Plant Research (IPK), 科学家近日在《The Plant Journal》上发表了一篇数字PCR方法高通量测定受精前大麦花粉核减数分裂重组率测量的相关文献,High-throughput measuring of meiotic recombination rates in barley pollen nuclei using Crystal Digital
PCR™
(原文链接:https://doi: 10.1111/tpj.15305)。采用Crystal Digital PCR™技术,在不进行全基因组扩增(WGA)的情况下,对大麦花粉核进行减数分裂重组率的高通量检测基因分型结果的分析,利用耐热限制性内切酶TaqI对花粉核进行预处理后
,单孔可以检测3000个花粉核,数小时即可完成检测。
实现更高的样本通量和进一步提高基因分型效率,检测了杂交植株中两个着丝点和两个远端染色体间隔的减数分裂重组率,检测 >42000单个花粉核(每株分析4900个核),获得的花粉核重组率与种群分离频率接近一致。

方法基于Crystal™Digital PCR技术,实现的单个大麦花粉核基因分型,提高了工作效率,在种内杂交植株的确定染色体间隔内进行可靠、快速和高通量的减数分裂重组率检测。同时不同核和基因组大小物种的核可成功包进微滴,如拟南芥(Arabidopsis thaliana),大豆(Glycine max),荷兰芹(Petroselinum crispum),甜椒(Capsicum annuum) ,青稞(Hordeum vulgare ) ,蚕豆( Vicia faba ),证明该方法广泛适用于植物的单核基因分型检测。
减数分裂作为有性生殖的基础,通过产生单倍体细胞和基于同源重组(HR)产生的遗传变异来实现(Mercier等人,2015;2018)。HR通过染色体交叉(CO)、同源染色体之间的联会,交换等来确保减数分裂染色体分离,同时保证遗传变异在育种过程中发挥作用。
在植物中,同源重组可以通过几种技术检测到(Lambing和Heckmann, 2018),例如通过减数分裂染色体分析进行细胞学检测(例如,Chelysheva等人,2012;Higgins等人,2012),通过测序进行基因分型和种群隔离的分子标记或荧光标记株系(FTLs)。FTLs在拟南芥中是检测花粉或种子中减数分裂重组事件的有效方法。但FTLs不适用于作物,因为作物基因组特别大,使用FTLs方法检测既费力又昂贵。此外,不同的作物或某些基因型不适合遗传转化。
作为替代方法,有文献报道使用小孢子(四分体或花粉核)基因分型或测序方法,直接检测减数分裂产物中减数分裂重组结果。但是作物小孢子的测序/基因分型相当昂贵,因此可以检测的数量有限,特别是对于谷物类大基因组物种。
在受精前检测雄配子的减数分裂重组率具备特定的优势:单个个体的数量大;分离群体的分子标记分析或耗时细胞观察染色体交叉(CO)独立;即时测量杂交配子中的染色体交叉CO频率,而不会产生分离失真。尽管在受精前直接测量配子重组事件具有优势,由于配子DNA含量有限,通常测序/基因分型方法需要进行全基因组扩增(WGA),链读测序。另一方面,直接通过PCR方法分析单个配子的基因分型方法也由于单倍体配子的低DNA含量,且无法实现高通量的基因分型检测。在拟南芥中有报道利用等位基因特异性PCR扩增杂交花粉DNA减数分裂重组频率可以识别在小的特定区域(通常在重组时为bp–kb)花粉分型。在大麦中,单花粉核基因分型是通过荧光细胞分选从种内杂交中分离出单个单倍体花粉核,然后进行WGA和多位点KASP基因分型或单细胞基因组测序完成的。单个单倍体花粉核的DNA有限,且WGA价格较高,导致分析样品的数量有限,无法进行高通量样本检测。
Crystal™数字PCR方法大麦单花粉核基因分型
为了突破受精前直接测定配子减数分裂重组率的局限性,
文章中探索了是否可以使用Crystal™数字PCR技术(naica®微滴芯片数字PCR系统, Stilla Technologies, 法国; Madic et al., 2016) (如图1)实现大麦单个
花粉核的快速基因分型,无需高成本、耗时的全基因组扩增(WGA)。
通过有限稀释和样本进行分区,即将单个花粉核包裹进微滴中,Crystal™数字PCR实现了高样本通量和低成本的花粉核的基因分型,我们成功地对两个着丝点和两个远端染色体间隔的减数分裂重组率进行测
定,后代植株与大麦单花粉核检测结果接近。
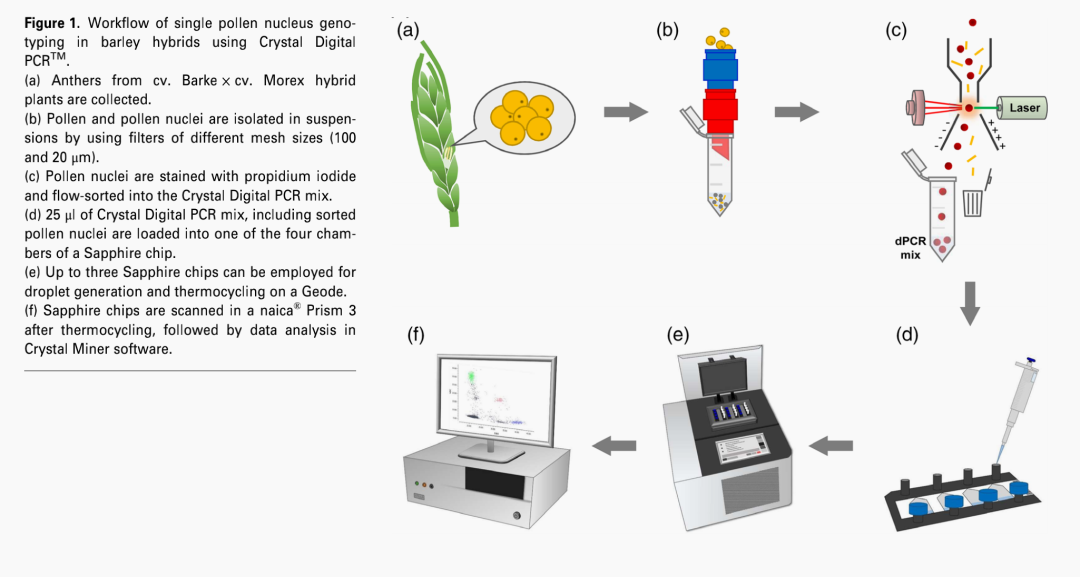
图1:Crystal™
数字PCR方法大麦单花粉核基因分型的工作流程
基于现有大麦基因组序列数据和已发表的多态性标记,比较cv. Morex 和cv. Barke后,在1号和3号染色体的4个染色体间隔(如图2)选择6个InDels和1个SNP,设计对应的基因多态性的寡核苷酸,扩增约100-200 bp的片段,以及区分亲本等位基因的双标记TaqMan探针(原文增补资料表S1和S2)。Barke特异性探针用HEX标记,Morex特异性探针用FAM标记,所有探针在3端用BHQ-1淬灭。
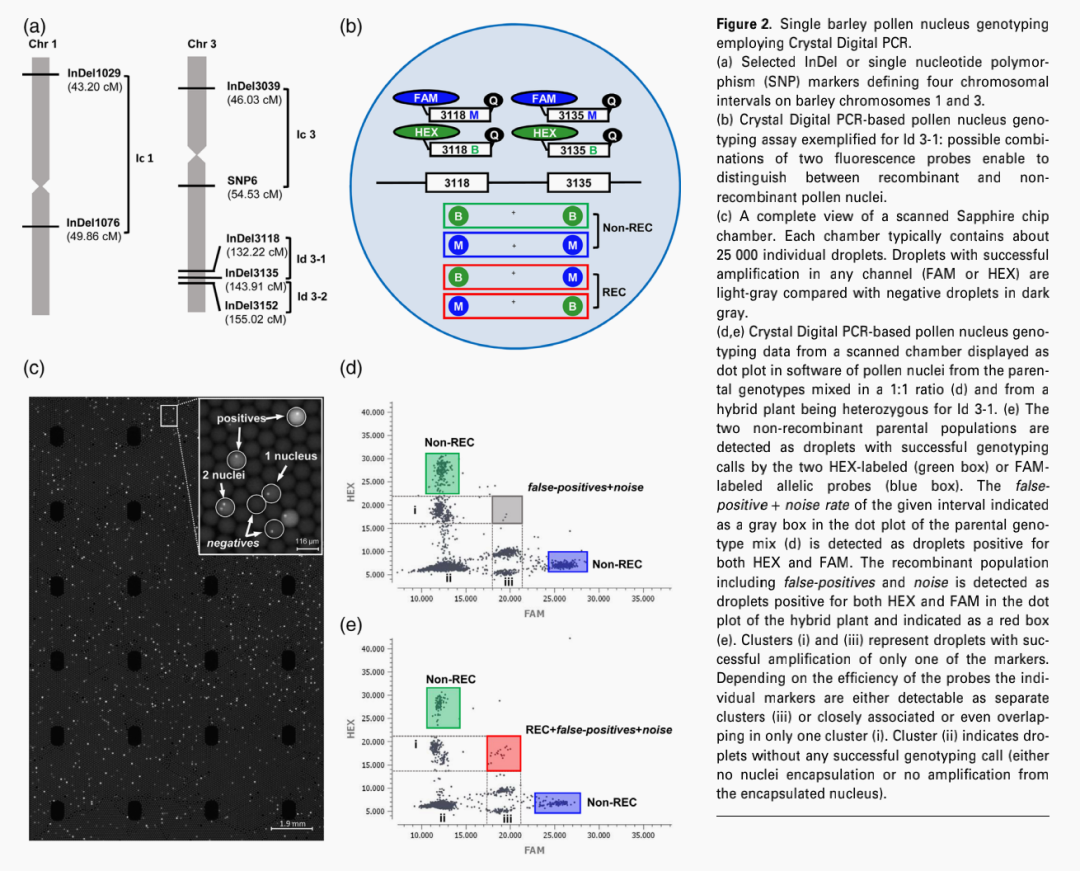
图2. Crysta
l
™
数字PCR方法大麦花粉单核基因分型设计
在进行花粉核减数分裂重组率检测时采用双重探针法,如前期可行性验证时检测的InDel3118和InDel3135之间的间隔Id 3-1,用HEX标记Barke (B)等位基因特异性探针(绿色),用FAM标记Morex (M)等位基因特异性探针(蓝色)(图2b),将来自亲本基因型的花粉核以1∶1的比例混合,同时也检测了Id 3-1杂合的杂交植物的花粉核。
在亲本混合样本检测中,两种亲本基因型的微滴相等,两种标记显示相同的荧光(B的HEX或M的FAM)(图2b)。
在杂交材料样本检测中下,预计会出现代表重组事件的不同微滴群,即同时显示两种颜色的微滴(InDel3118为HEX,InDel3135为FAM)(图2b)。
在实验过程中发现,亲代基因型得到了数量大致相等的微滴,它们对两种标记物显示出相同的荧光(图2d,e,绿色和蓝色矩形)。
在对杂交植物的花粉核的检测中,检测到具有两种颜色(HEX和FAM)的微滴,表明重组事件(图2e,红色矩形)。
此外,可以区分只有一个标记成功扩增的微滴(图2d,e,簇i和iii)以及没有任何扩增的微滴(图2d,e,簇ii),证明使用Crystal
™
数字PCR方法对单个花粉核进行包裹和基因分型是完全可行的。
Crystal™数字PCR系统具有极高的分辨率,因此在那些成功扩增标记物的微滴中,也可以观察到微滴内的细胞核(图2c),研究者通过对微滴包裹核的数量分析进一步优化实验,通过用热稳定的限制性酶预处理花粉核来提高基因分型的效率,且因为细胞核数量与单个包裹细胞核的微滴数量呈正相关,提出上样细胞核的最佳数量范围(因不同物种细胞核大小不同而有所不同)。
采用naica@微滴芯片数字PCR即Crystal™
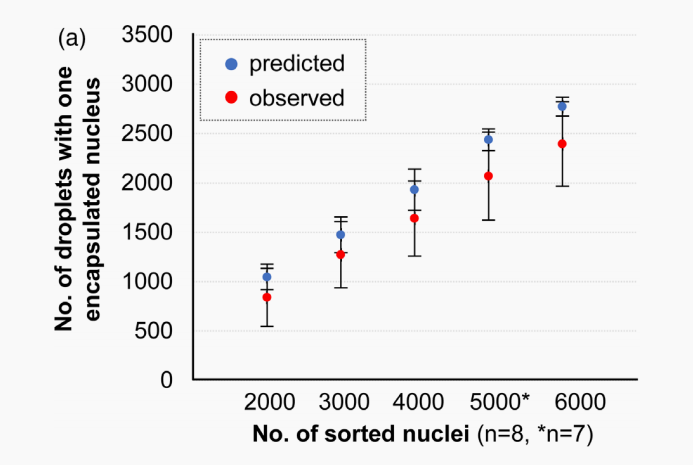
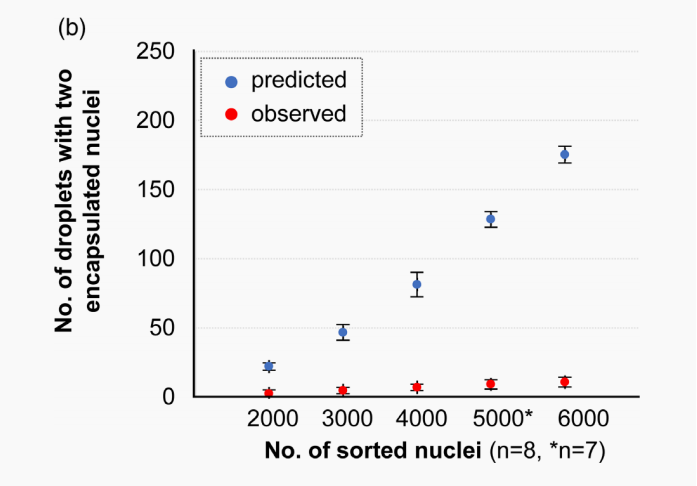
数字PCR方法可以评估和包被经流式分选PI染色的花粉核,每个反应孔可以可靠地评估多达6000个核,对不同数量的花粉核(2000-6000)的包裹率结果显示,核的数量与具有单个包裹核的微滴数量呈正相关(图3a),约有一半被封装(39.9-42.4%),接近泊松分布的预测值。含有一个以上核的微滴数量,如含两个核,也与分选核的数量正相关,但从计算的泊松分布来看,这一数值与预测值比较要低于得多(图3b)。当来自双亲基因型的核被包裹在一个微滴时,这些微滴可能导致假阳性,因此在进一步的实验中,我们从计算中排除了所有含有大于一个核的微滴。
通过综合研究可被微滴包裹的核数、包裹一个以上核的微滴百分比,PCR效率,单个大麦穗可获得的平均花粉核数(通常是每个穗3万至5万个可流式分选的花粉核),本研究确定在后续所有分析中使用每个反应孔中的3000个分选花粉核作为基准。
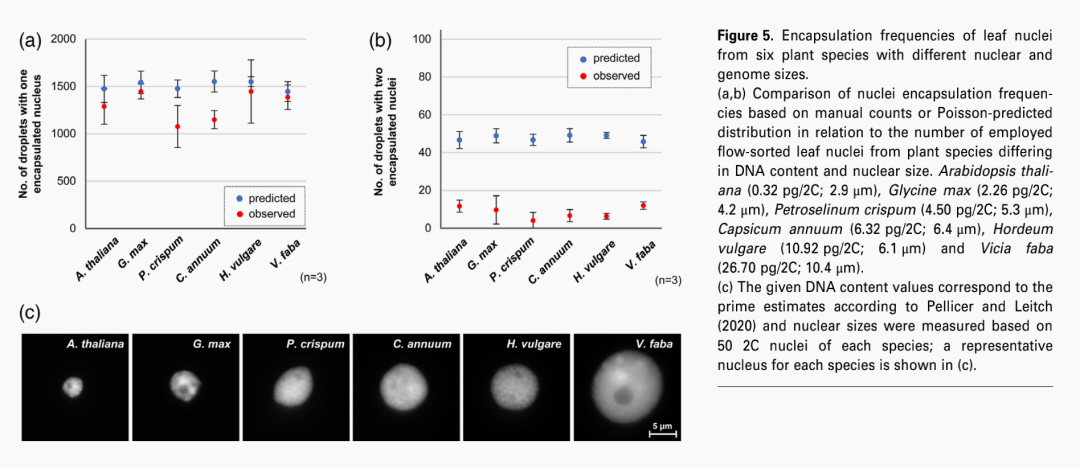
评估核大小和包裹多于一个核对于微滴包裹效率的影响,选取3000个不同DNA含量和核大小的六种植物叶片核进行微滴包裹验:拟南芥(Arabidopsis thaliana)0.32 pg/2C;2.9 μm,大豆(Glycine max)2.26 pg/2C;4.2 μm,荷兰芹(Petroselinum crispum)4.50 pg/2C;5.3 μm,甜椒(Capsicum annuum)6.32 pg/2C;6.4 μm ,青稞(Hordeum vulgare ) 10.92 pg/2C;6.1 μm,蚕豆( Vicia faba )26.70 pg/2C;10.4 μm。
图4:不同核和基因组大小的六种植物叶片核的微滴包裹效率
DNA值参考Pellicer和Leitch(2020)进行估算,核大小是通过每个物种在荧光显微镜下,测量50个经DAPI染色的核而确定(图4c)。在所有物种中,花粉核的包裹效率与大麦花粉核的包裹效率接近一致,接近泊松分布预测值(图4a),而多个核的包裹效率远低于预期值(图4b)。
为了评估测量到的花粉核中的雄性减数分裂重组率是否反映了植物中的重组率,对分离群体中的子代植物的所有4个间隔进行了基因分型。花粉核样品中观察到接近一致的重组率,假设雄性和雌性配子的贡献相等:Ic1 (n = 99)的8.6%;Ic3 (n = 99株)占7.6%;Id 3-1为11.1% (n = 54个子代);而Id 3-2为12.2% (n = 98个子代植株)。
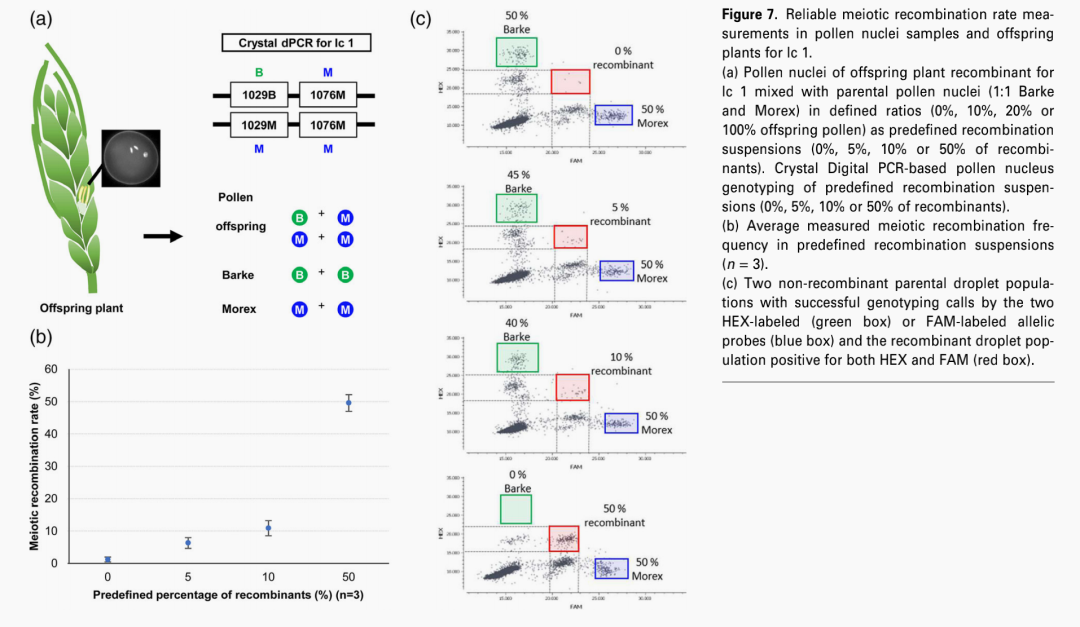
图5:可靠测定花粉核样品和子代植物中Ic1减数分裂重组率
为了进一步证明我们的基因分型的可靠性,选择Ic1重组子代植株。重组植株的花粉核以不同的确定比例(0%、10%、20%、100%)与亲本Barke和Morex花粉核的1:1比例(100%、90%、80%、0%非重组花粉核)混合,测定Ic 1的重组率,如图5。
使用混合的具有确定数量的重组花粉核和非重组花粉核的样本,并没有偏离预期的重组率,证实了Crystal™数字PCR方法大麦单花粉核减数分离重组率的稳健性。在Id 3-1和Ic 3中也成功测量了混合样本的重组率。
Crystal™ 数字PCR技术检测花粉核中减数分裂重组的可靠性和生物学意义
通过Crystal™ 数字PCR技术测量了两条独立染色体的远端或中间位置的四个选定染色体间隔内减数分裂重组率。在校正间隔特异性噪声后,通过上述基因分型方法,花粉核中雄性减数分裂重组率的平均值分别为:7.1±0.9%(Ic 1);6.0±3.1%(Ic 3);12.0±2.2%(Id 3-1);10.9±1.7%(Id 3-2),这与2015年Zhou et al.对于Barke × Morex的雄性和雌性减数分裂重组报道值接近一致, Ic 1,6.7 cM;Ic 3 ,8.5 cM;Id 3-1,11.7 cM,;Id 3-2 ,11.1 cM。减数分裂重组率间微小差异,例如Ic 3的6.0±3.1%和8.5 cM,可能是基于植物不同的栽培条件(已知的减数分裂重组率影响因素,Dreissig等,2019;Francis等,2007;Higgins等,2012;Modliszewski等,2018;Zhang et al.,2017)。另外,本实验测量的为雄性配子重组频率,重组率的性别特异性差异(Phillips等,2015)也可能是微小差异的原因。Ic 1植株间的重组率较为一致。Ic 3和Id 3-1的重组率在植株间差异较大,可能某些遗传间隔在减数分裂重组更具可变性。
当检测不同比例的重组混合样本的重组率时,子代植物的花粉核与Barke和Morex花粉核以不同的比例混合,通过基于Crystal™ 数字PCR的方法测量的重组率与混合样本的预期重组率相匹配。因此,Crystal™ 数字PCR技术检测花粉核的基因型方法可以可靠地测量特定花粉核重组频率。此外,在植物中发现的重组率,ic1的重组率为8.6%,Ic 3的重组率为7.6%,Id 3-1的重组率为11.1%,Id 3-2的重组率为12.2%,表明花粉核内的雄性重组率和雌性重组率一致。
综上所述:基于Crystal Digital PCR的单花粉核基因分型测量方法,在数小时内即可实现可靠地、高通量的测量花粉核中的减数分裂重组率,与大麦种群隔离中描述和发现的比率接近一致。同时发现具有不同细胞核和基因组大小的物种的细胞核的成功包裹表明,所提出的方法广泛适用于多种植物的单个细胞核的基因分型。
法国Stilla Technologies公司naica®六通道数字PCR系统,源于Crystal微滴芯片式数字PCR技术,自动化微滴生成和扩增,每个样本孔可实现6荧光通道的检测,智能化识别微滴并进行质控,3小时内即可获得至少6个靶标基因的绝对拷贝数浓度。

为了方便大家更好地学习数字PCR相关知识,我们为大家提供了Gene-π数字PCR学堂,您可以选择感兴趣的应用教程,其中包含从实验设计到数据分析的详细工作流程。
您还可以通过搜索导航工具直接搜索特定的项目("dPCR的DNA准备","荧光溢出"等),或点击我们的"HOW TO" 选择您需要的部分(设计、分析、报告)。
详细信息可登录北京深蓝云生物科技有限公司网站查看。



















 个人中心
个人中心
 我是园区
我是园区




